El aprendizaje profundo sueña con nuevas estructuras de proteínas
Los investigadores demuestran que una red neuronal entrenada exclusivamente para predecir las formas de las proteínas también puede generar otras nuevas
Al igual que se pueden crear imágenes convincentes de gatos mediante la inteligencia artificial, ahora se pueden fabricar nuevas proteínas con herramientas similares. En un artículo publicado en Nature, un equipo formado por investigadores de la Universidad de Washington, el Instituto Politécnico Rensselaer y la Universidad de Harvard describe el desarrollo de una red neuronal que "alucina" proteínas con estructuras nuevas y estables.
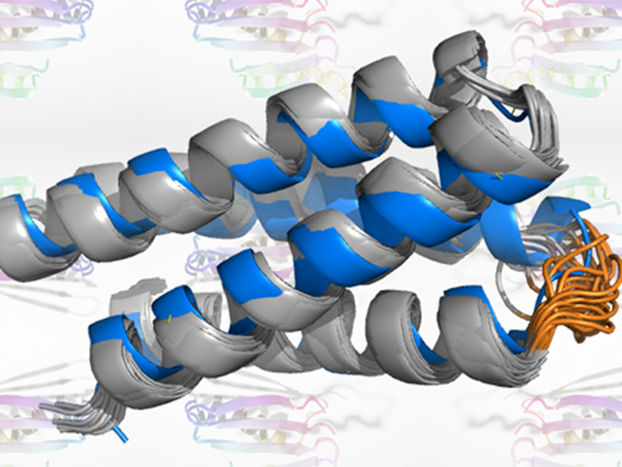
Una red neuronal "alucinó" con las proteínas que se sintetizaron para confirmar su estructura.
Rensselaer Polytechnic Institute
"El potencial para alucinar proteínas totalmente nuevas que se unen a determinadas biomoléculas o forman sitios activos enzimáticos deseados es muy emocionante", dijo Gaetano Montelione, profesor de química y biología química en Rensselaer, donde se analizaron versiones sintetizadas de proteínas "alucinadas" inventadas por una red neuronal.
Las proteínas son moléculas similares a cuerdas que se encuentran en todas las células y que se pliegan espontáneamente en intrincadas formas tridimensionales. Estas formas plegadas son clave en casi todos los procesos de la biología, como el desarrollo celular, la reparación del ADN y el metabolismo. Pero la complejidad de las formas de las proteínas dificulta su estudio. Los bioquímicos suelen utilizar ordenadores para predecir cómo se pueden plegar las cadenas o secuencias de proteínas. En los últimos años, técnicas de inteligencia artificial como las redes neuronales y el aprendizaje profundo han revolucionado la precisión de este trabajo.
"Para este proyecto, creamos secuencias de proteínas completamente aleatorias e introdujimos mutaciones en ellas hasta que nuestra red neuronal predijo que se plegarían en estructuras estables", dijo el coautor principal Ivan Anishchenko, un becario postdoctoral en el laboratorio de Baker en el Instituto de Diseño de Proteínas de la Facultad de Medicina de la Universidad de Washington. "En ningún momento guiamos el software hacia un resultado concreto: estas nuevas proteínas son simplemente lo que el ordenador sueña".
En el futuro, el equipo cree que debería ser posible dirigir la inteligencia artificial para que genere nuevas proteínas con características útiles. "Nos gustaría utilizar el aprendizaje profundo para diseñar proteínas con función, incluyendo fármacos basados en proteínas, enzimas, lo que sea", dijo el coautor principal Sam Pellock, un becario postdoctoral en el laboratorio de Baker.
El equipo de investigación generó 2.000 nuevas secuencias de proteínas que se predijo que se plegarían. Más de 100 de ellas se produjeron en el laboratorio y se estudiaron. El análisis detallado de tres de estas proteínas confirmó que las formas predichas por el ordenador se hicieron realidad en el laboratorio.
"Nuestros estudios de RMN en solución, junto con las estructuras cristalinas de rayos X determinadas por el equipo de la Universidad de Washington, demuestran la notable exactitud de los diseños de proteínas creados por el método de alucinación", afirmó la coautora Theresa Ramelot, investigadora principal del laboratorio de Montelione en el Centro Rensselaer de Biotecnología y Estudios Interdisciplinarios.
Montelione señala: "El enfoque de alucinación se basa en observaciones anteriores que realizamos junto con el laboratorio Baker y que revelan que la predicción de la estructura de las proteínas con el aprendizaje profundo puede ser bastante precisa incluso para una sola secuencia de proteínas, sin recurrir a las predicciones de contacto que suelen obtenerse mediante el análisis de muchas secuencias de proteínas relacionadas con la evolución."
"Este enfoque simplifica en gran medida el diseño de proteínas", dijo el autor principal David Baker, galardonado con el Premio Breakthrough 2021 en Ciencias de la Vida. "Antes, para crear una nueva proteína con una forma particular, la gente primero estudiaba cuidadosamente las estructuras relacionadas en la naturaleza para llegar a un conjunto de reglas que luego se aplicaban en el proceso de diseño. Se necesitaban nuevos conjuntos de reglas para cada nuevo tipo de pliegue. Aquí, al utilizar una red de aprendizaje profundo que ya capta los principios generales de la estructura de las proteínas, eliminamos la necesidad de reglas específicas de los pliegues y abrimos la posibilidad de centrarnos directamente solo en las partes funcionales de una proteína."
"Explorar cómo utilizar mejor esta estrategia para aplicaciones específicas es ahora un área activa de investigación, y es aquí donde espero que se produzcan los próximos avances", dijo Baker.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.