La enzima silencia las secuencias similares a las del virus
La metiltransferasa Dnmt1 no sólo es responsable del mantenimiento
A menudo, la biología no encaja en categorías claras. En una publicación reciente, una enzima a la que los investigadores habían asignado previamente una tarea distinta se sale de su caja. No sólo mantiene el estado epigenético del genoma, sino que puede silenciar específicamente otros segmentos de ADN con características similares a las de los virus.
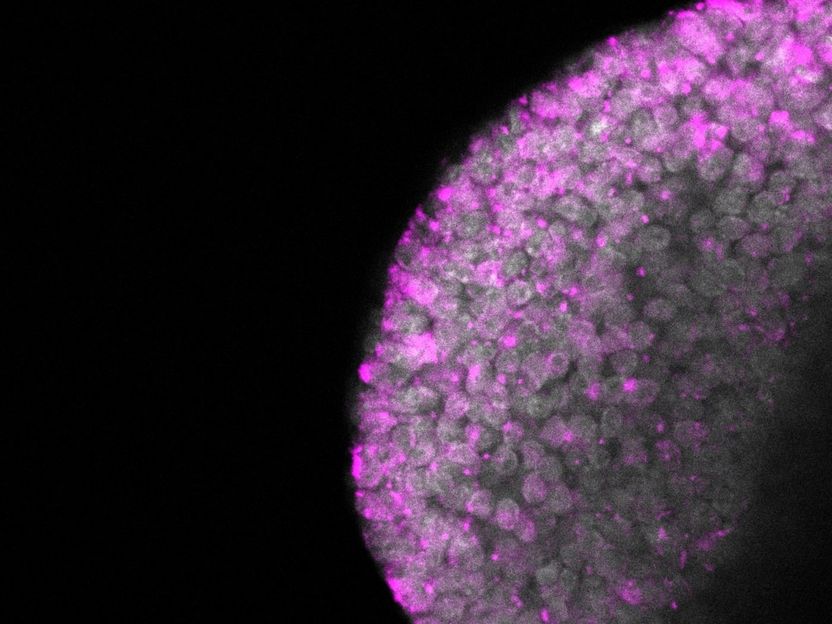
Después de la eliminación de la metiltransferasa Dnmt1 en un cigoto, los retrotransposones IAP están regulados (moléculas de retrotransposones marcadas en rosa)
Abhishek Sampath Kumar, MPI f. mol. Genet
Es la sección prohibida en la biblioteca de la vida: Algunas partes del ADN contienen información perjudicial. Las enzimas metiltransferasas dotan a estas secciones de etiquetas químicas, haciendo que la célula las ignore. Al igual que el bibliotecario pone etiquetas de advertencia en ciertos libros, estas enzimas dejan intacta la secuencia genética añadiendo pequeños paquetes de carbono (grupos metilo) a la molécula de ADN, lo que significa que están operando epigenéticamente.
Durante décadas, parecía haber una clara división del trabajo: las metiltransferasas Dnmt3a y Dnmt3b buscan segmentos adecuados de ADN y los adornan con grupos metilo. A continuación, la enzima hermana Dnmt1 se limita a copiar las etiquetas cuando sólo un lado de la cadena de ADN está metilado tras la división celular o cuando se ha perdido la mitad de las etiquetas de las dos cadenas.
Sin embargo, no es tan sencillo, como demuestra el grupo de investigación de Alexander Meissner en el Instituto Max Planck de Genética Molecular en un nuevo artículo publicado en Nature Structural and Molecular Biology. Según el nuevo estudio, la Dnmt1 tiene, en contra de lo que se suponía hasta ahora, la capacidad de dirigirse a secuencias específicas del genoma del ratón y fijarles marcas de metilo fuera de sus tareas de mantenimiento.
El óvulo fecundado pierde su metilación hasta que el embrión temprano se convierte en un estado similar a una burbuja, el blastocisto. "El blastocisto tiene uno de los niveles generales más bajos de metilación del ADN durante el desarrollo del ratón, pero apenas 72 horas después, el genoma ha pasado a un estado altamente metilado", dice Meissner. "Por eso nos fijamos en estos puntos temporales especialmente interesantes".
¿Pero por qué es necesario desactivar segmentos específicos del ADN en primer lugar? Las células utilizan la metilación para determinar qué programas genéticos deben ejecutar. Pero hay una razón más insidiosa, explica Meissner: "La mayoría de los genomas -incluidos los de humanos y ratones- contienen miles de secuencias derivadas de virus que se han incrustado en los genomas de sus huéspedes durante los últimos millones de años".
Las secuencias similares a los virus son transposones, también conocidos como "genes saltarines". Tienen la capacidad de replicarse espontáneamente e integrarse en cualquier lugar del ADN. Los transposones se han extendido por todo el genoma y constituyen aproximadamente el 40% del genoma en ratones y humanos.
"La metilación ayuda a mantener bajo control estos tramos del genoma potencialmente dañinos", afirma Chuck Haggerty, candidato al doctorado y uno de los primeros autores de la publicación. "Si un transposón o un virus salta al centro de un gen, podría alterar su función. Y si se permitiera que los transposones se extendieran sin control por el genoma, su integridad podría estar en peligro".
"Nos dimos cuenta de que el genoma de los embriones seguía ganando metilación incluso cuando eliminábamos Dnmt3a y 3b", dice Haggerty. Los investigadores modelaron esto en un cultivo celular, eliminaron de nuevo Dnmt3a y 3b, y luego desactivaron adicionalmente Dnmt1. La metilación se detuvo en este punto y, tras un breve periodo de tiempo, el genoma estaba casi completamente desmetilado.
"Curiosamente, cuando se volvió a encender sólo la Dnmt1, los niveles de metilación empezaron a aumentar a pesar de que no había marcas de metilación previas que mantener", dice Haggerty. Esto fue una confirmación concluyente de que la enzima Dnmt1 puede metilar el ADN de nuevo, o "de novo".
El análisis de la secuencia mostró que la Dnmt1 muestra una especial preferencia por las regiones de ADN que contienen un determinado tipo de transposón, que es común entre los roedores. Las "Partículas Intracisternas A" (IAP) son retrotransposones cuyo ciclo vital es sorprendentemente similar al de los retrovirus, como el virus de la inmunodeficiencia humana (VIH).
La célula lee la secuencia retroviral de su ADN y produce moléculas de ARN que contienen el plano de las proteínas. Entre ellas hay una enzima que convierte el ARN en ADN y otra que inserta este ADN en el genoma de la célula.
De hecho, la Dnmt1 se dirige de forma muy específica a las secuencias de PAI en el genoma de las células de ratón, afirma Haggerty: "Metiló algunas secciones hasta un 18 por ciento, un nivel muy alto para una enzima que no se esperaba que tuviera esa capacidad".
Los investigadores ya han descubierto algunas pistas sobre el modo en que la Dnmt1 busca su objetivo. En experimentos basados en cultivos celulares, identificaron otras proteínas que parecen unirse a la enzima Dnmt1 y que se sabe que interactúan con los retrotransposones.
Al estudiar los retrotransposones, los científicos alcanzaron rápidamente los límites de la tecnología convencional. "Examinamos fragmentos de ADN relativamente cortos en la secuenciación tradicional", dice la científica computacional Helene Kretzmer, también primera autora del trabajo. Buscamos solapamientos y similitudes en las secuencias y las unimos". Dado que las copias de retrotransposones que están dispersas por el genoma comparten una secuencia similar, a menudo es difícil saber exactamente qué región estamos viendo."
En consecuencia, los investigadores utilizaron un método de secuenciación en el que la cadena de ADN se corta en segmentos relativamente largos que luego se hacen pasar por poros de tamaño nanométrico en un chip. "Los métodos convencionales nos dan fragmentos de unas 150 a 300 bases, pero la secuenciación por nanoporos abarca todo el transposón con fragmentos derivados de 10.000 a 30.000 bases", dice Kretzmer. "Para el análisis de la metilación de secuencias repetitivas, este método no tiene actualmente parangón".
"Por supuesto, la principal tarea de Dnmt1 sigue siendo, sin duda, mantener la metilación", dice Meissner. "Si se observa todo el genoma, la actividad global de novo de la enzima es baja, pero hemos demostrado que en contextos específicos puede ser más significativa".
Recientemente, otros grupos de investigación habían encontrado cada vez más pruebas de que la Dnmt1 es capaz de metilar el ADN por sí misma en una pequeña medida. El estudio actual corrobora esta sospecha. "Hemos puesto fin a años de debate y demostramos que tal vez haya que revisar algunas clasificaciones de las metiltransferasas del ADN", afirma Meissner. "Al mismo tiempo, abrimos un nuevo capítulo al preguntar: ¿en qué otros contextos podría tener relevancia biológica la actividad de novo de Dnmt1?".
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Haggerty C, Kretzmer H, Riemenschneider C, Kumar AS, Mattei AL, Bailly N, Gottfreund J, Giesselmann P, Weigert R, Brändl B, Giehr P, Buschow R, Galonska C, von Meyenn F, Pappalardi MB, McCabe MT, Wittler L, Giesecke-Thiel C, Mielke T, Meierhofer D, Timmermann B, Müller FJ, Walter J, Meissner A; "Dnmt1 has de novo activity targeted to transposable elements"; Nat Struct Mol Biol.; 2021 Jun 17.