Bloqueo definido: Fotoencapsulado enzimático para el estudio de la regulación de los genes a través de la metilación del ADN
Anuncios
La adición y eliminación de grupos metilo en el ADN desempeña un papel importante en la regulación de los genes. A fin de estudiar estos mecanismos con mayor precisión, un equipo alemán ha desarrollado un nuevo método mediante el cual se pueden bloquear y luego desbloquear sitios específicos de metilación en un momento preciso mediante la irradiación con luz (fotoenjaulado). Como se informa en la revista Angewandte Chemie, el regente requerido se produce enzimáticamente, in situ.
© Wiley-VCH
Aunque se ven muy diferentes y sirven para funciones completamente diferentes, todas las células de nuestro cuerpo tienen idéntico ADN. Sin embargo, no utilizan los mismos genes. Algunos genes se activan y otros se desactivan, dependiendo del tipo de célula y del momento en el tiempo. Los "interruptores" son cambios químicos en los bloques de construcción del ADN. Estos cambios se llaman modificaciones epigenéticas. Un mecanismo de regulación importante es la metilación y desmetilación, es decir, la unión y la eliminación de un grupo metilo (-CH3). Los patrones de metilación de las células cancerosas, por ejemplo, difieren de las células sanas. Durante la metilación, las enzimas conocidas como metiltransferasas (MTases) transfieren un grupo metilo del S-adenosil-ʟ-metionina(AdoMet) a la molécula objetivo.
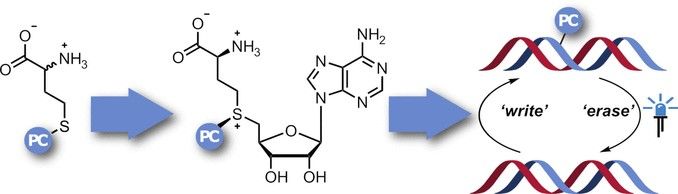
A fin de estudiar más detenidamente el propósito y la función de esta regulación y determinar las pautas de metilación, sería útil disponer de "instrumentos" para inhibir específicamente la metilación en lugares determinados y luego levantar la inhibición en un momento definido. Con este fin, un equipo dirigido por Andrea Rentmeister eligió utilizar un método conocido como fotoenjaulado. En este método, una "fotojaula" es una molécula que se deshace al ser irradiada, como un grupo de 2-nitrobencilos. La jaula primero bloquea la ubicación del objetivo, luego la irradiación dirigida con luz actúa como un "interruptor" para eliminar el bloqueo.
La idea era equipar a los análogos de AdoMet con una fotocaja que luego se transfiere a los sitios de metilación. Sin embargo, los análogos del AdoMet se descomponen en soluciones acuosas y no pueden entrar en las células. Por lo tanto, el equipo de la Universidad de Münster quería producirlos in situ. En el cuerpo, el AdoMet se produce a partir del aminoácido metionina a través de la acción de la enzima metionina adenosil transferasa (MAT). La síntesis de los análogos del AdoMet requiere metionina con una fotocaja de nitrobencilo adjunta y una MAT que pueda utilizar un sustrato tan alterado. Comenzando con una enzima MAT de un organismo unicelular(Cryptosporidium hominis), los investigadores pudieron cambiar cuidadosamente los aminoácidos específicos de la enzima para aumentar el tamaño de su cavidad de unión hidrofóbica para que pudiera contener el grupo nitrobencilo. Un análisis de la estructura cristalina mostró que el análogo de ADoMet está ligado en la cavidad de este fotoencapsulado MAT (PC-MAT). Basándose en esta información, el equipo también produjo un segundo PC-MAT basado en una enzima MAT termoestable de la arquebacteria Methanocaldococcus jannaschii.
Ambos PC-MAT son compatibles con las MTasas de ADN y ARN y permitieron adjuntar fotocajas a todos los sitios de metilación natural del ADN de un plásmido. La irradiación con luz eliminó el bloqueo.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.