Más flexible de lo previsto
Nuevos hallazgos sobre la proteína de punta del SARS-CoV-2
Para combatir el coronavirus SARS-CoV-2, se han intensificado las investigaciones en busca de nuevas vacunas y tratamientos. Para entrar en las células, el virus necesita la proteína de punta en su superficie. Los investigadores del Instituto Paul-Ehrlich, en cooperación con algunos grupos de investigación de Alemania (EMBL en Heidelberg, MPI de Biofísica en Frankfurt) han analizado la proteína espiga en su entorno natural utilizando imágenes de alta resolución y métodos asistidos por ordenador. Al hacerlo, han descubierto un grado de movilidad de los picos en la superficie del virus que no se esperaba anteriormente. Los resultados se publican en Science en su edición online del 18 de agosto de 2020
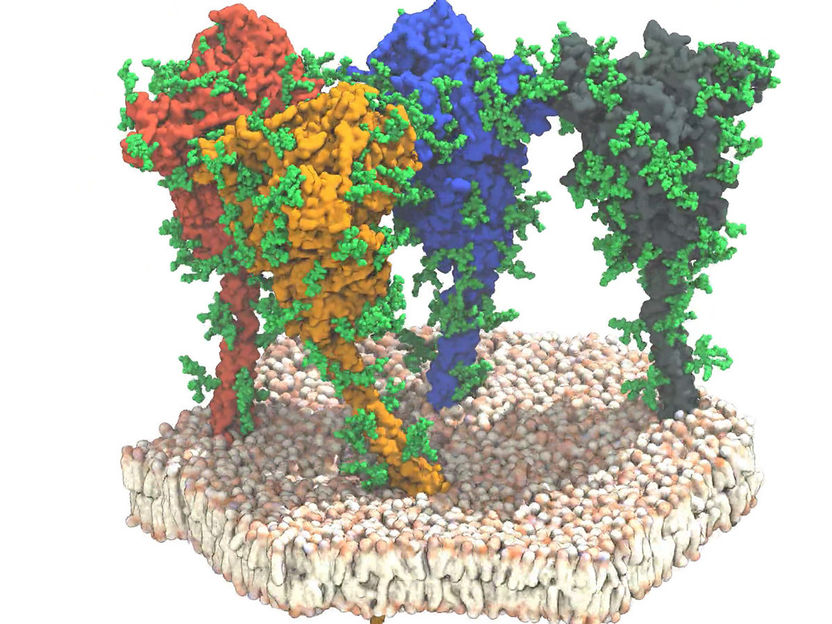
Simulación de cuatro proteínas de pico (rojo, naranja, azul y gris) del virus del SARS-CoV-2. Las proteínas y los lípidos se muestran en la representación de la superficie. Las cadenas glicánicas protectoras se muestran en verde.
MPI for Biophysics, Frankfurt/Main
El coronavirus SARS-CoV-2 necesita las proteínas de punta (S) de su superficie para unirse a receptores específicos en la superficie de las células humanas. Utiliza esta vía para infectar las células. Estas estructuras en forma de espiga están en el centro del desarrollo de la vacuna, ya que su papel es actuar como antígeno para inducir una respuesta inmunológica en los humanos que los proteja de COVID-19. Los investigadores están estudiando el virus intensamente, en particular, su estructura superficial, para comprender tanto las vacunas como el desarrollo de terapias eficaces para el tratamiento de los pacientes infectados. En este contexto, el conocimiento de las estructuras espaciales juega un papel importante, que son importantes para diversos procesos como la entrada en las células diana.
La combinación de las más modernas tecnologías de crio-tomografía electrónica, promediación de sub-tomogramas y simulación de dinámica molecular permite el análisis estructural de las estructuras moleculares en su entorno natural a una resolución casi atómica. La profesora Jacomine Krijnse Locker, jefa del grupo de investigación Loewe "Microscopía electrónica de patógenos" y su equipo de investigación en el Paul-Ehrlich-Institut, en cooperación con la sección del Paul-Ehrlich-Institut que dirige el profesor Michael Mühlebach, e investigadores del Laboratorio Europeo de Biología Molecular (EMBL), el Max-Planck-Institut for Biophysics y el Instituto de Biofísica de la Universidad Goethe de Francfort/Main han analizado la proteína de espiga en su entorno natural mediante métodos de imagen y modelos de simulación. Para ello, utilizaron partículas de SARS-CoV-2 extraídas del sobrenadante de células infectadas. Con la ayuda de los más modernos métodos de microscopía electrónica en el EMBL, se generaron 266 imágenes de criografia de alrededor de 1000 virus diferentes, cada uno con 40 picos en sus superficies en promedio. Utilizando el promedio de la subtomografía y el procesamiento de imágenes, se pudo obtener información importante sobre las estructuras de un total de 40.000 picos.
El resultado es favorable para el desarrollo de una vacuna. La parte superior en forma de esfera o en forma de V de la espiga, en condiciones naturales, muestra una estructura que puede ser fácilmente reproducida por proteínas recombinantes utilizables para el desarrollo de una vacuna. Sin embargo, los hallazgos en el eje utilizado para fijar la parte esférica de la espiga a la superficie del virus eran nuevos.
Poco se sabía anteriormente sobre esta estructura. Ahora, los investigadores han descubierto que el eje es extremadamente flexible. Las imágenes mostraron que rara vez era perpendicular a la membrana, sino que se inclinaba en todas las direcciones.
El equipo identificó cuatro dominios diferentes (secciones) en el eje, que llamaron el dominio de la "cadera", la "rodilla" y el "tobillo", y finalmente el dominio del pie, que está incrustado en la membrana. Utilizando una combinación de simulaciones dinámicas moleculares y criotomografía, los investigadores proporcionaron pruebas de que estos dominios pueden ejecutar movimientos de flexión. Los datos muestran que la parte esférica de los picos, que contiene el área de unión del receptor y los componentes necesarios para la célula objetivo, está conectada a un eje flexible. "Como un globo en una cuerda, los picos parecen moverse en la superficie del virus, lo que les permite encontrar el receptor para adherirse a la célula objetivo", como describe el profesor Jacomine Krijnes Locker los resultados. Finalmente, los análisis muestran que el eje está equipado con muchas cadenas de glicanos. Esto podría proporcionar al eje un manto protector, que lo protegerá de los anticuerpos neutralizantes. Esto puede ser aclarado en futuros experimentos y contribuirá a la comprensión de las propiedades inmunológicas de las proteínas de punta en el camino hacia vacunas efectivas.
Nota: Este artículo ha sido traducido utilizando un sistema informático sin intervención humana. LUMITOS ofrece estas traducciones automáticas para presentar una gama más amplia de noticias de actualidad. Como este artículo ha sido traducido con traducción automática, es posible que contenga errores de vocabulario, sintaxis o gramática. El artículo original en Inglés se puede encontrar aquí.
Publicación original
Turoňová* B, Sikora* M, Schürmann* C, Hagen WJH, Welsch S, Blanc FEC, Sören von Bülow S, Gecht M, Bagola K, Hörner C, v Zandbergen G, Mosalaganti S, Schwarz A, Covino R, Mühlebach MD, Hummer G, Krijnse Locker J, Beck M; "In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges"; Science; 2020 (*contributed in equal parts)