Rationelles Arbeiten auf kleinem Raum: Neue Einblicke in die Architektur zellulärer Proteinfabriken
Um Proteine zu erzeugen, besitzt jede Zelle ihren eigenen Maschinenpark: Die Ribosomen. Als Baupläne dienen dabei Botenmoleküle aus dem Zellkern. Meist schließen sich mehrere Ribosomen zu einem Polysom zusammen. Wissenschaftlern des Max-Planck-Instituts für Biochemie gelang es nun erstmals, die dreidimensionale Struktur von Polysomen aufzuklären: Sie sind so angeordnet, dass die erzeugten Proteine den größtmöglichen Abstand wahren, um sich nicht zu verheddern. Bisher glaubte man, Fehlfaltungen würden nur durch spezialisierte Proteine verhindert. Zudem ermöglicht die räumliche Struktur, dass das Botenmolekül geschützt und auf kurzem Weg weiter gereicht wird.
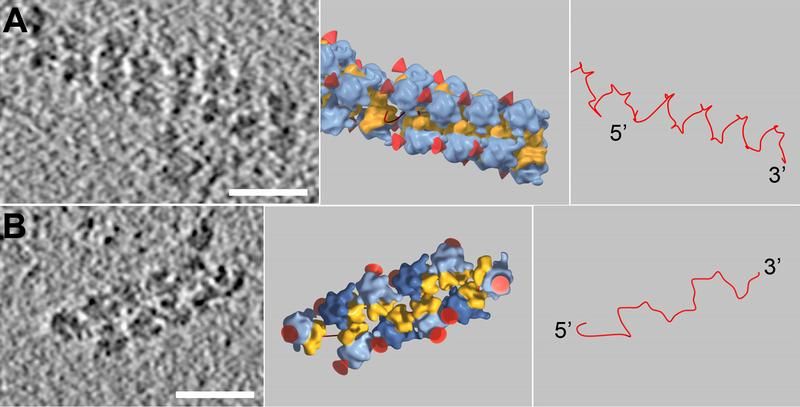
Die Abbildung zeigt die kryo-elektronentomographische Aufnahmen zweier Polysomen (links), die schematische Darstellung ihrer Struktur (mitte) sowie den Weg des Botenmoleküls (der mRNA) innerhalb des Polysoms (rechts). Die kleinen Untereinheiten der Ribosomen (gelb) sind ins Innere des Polysoms gerichtet und einander zugewandt, die großen Untereinheiten (blau) und die entstehenden Proteine (symbolisiert durch rote Kegel) orientieren sich nach außen. Eine regelmäßige "Kopf-an-Kopf"-Anordnung der Ribosomen führt zu einem schraubenförmigen Polysom (Abb. A, mitte), bei abwechselnder Anordnung erscheinen Polysomen in ebenflächiger Form (Abb. B, mitte). In beiden Fällen wird die mRNA auf möglichst direktem Weg von einem benachbarten Ribosom zum nächsten geschleust (Abb. A und B, rechts).
Florian Brandt, Max-Planck-Institut für Biochemie
In jedem Protein reihen sich mehrere Hundert oder Tausend Aminosäuren aneinander, die in den Ribosomen in einer bestimmten Reihenfolge verknüpft werden. Die Bauanleitung für die einzelnen Proteine bringt ein fadenförmiges Botenmolekül, die mRNA (messenger RNA) von der DNA im Zellkern zu den Ribosomen. Diese tasten die mRNA Schritt für Schritt ab und übersetzen die genetische Information in die Aminosäure-Sequenz der Proteine: Ist ein Abschnitt abgelesen und die entsprechende Aminosäure in die Kette eingefügt, rückt das Botenmolekül weiter und die nächste Aminosäure folgt. Damit der Vorgang besonders effizient abläuft und aus einer mRNA zahlreiche Proteine erzeugt werden, reihen sich in der Zelle mehrere Ribosomen zu einem sogenannten Polysom aneinander: Im Polysom läuft die mRNA wie am Fließband von einem Ribosom zum nächsten, sodass mehrere Proteine gleichzeitig entstehen. Wie diese Ribosomen-Kette im Detail aussieht, war bisher noch nicht bekannt.
Ein Kooperationsprojekt unter der gemeinsamen Leitung von Wolfgang Baumeister, Direktor der Abteilung Molekulare Strahlenbiologie, und Ulrich Hartl, Direktor der Abteilung Zelluläre Biochemie, nutzten nun die in Martinsried entwickelte Technik der Kryo-Elektronentomographie, um die Architektur dieser molekularen Fabriken sichtbar zu machen.
Die Kryo-Elektronentomographie ist ein Verfahren mit dem dreidimensionale Strukturen der Zelle direkt untersucht werden können. Der Trick: Die gesamte Zelle bzw. einzelne Zellbestandteile werden blitzartig "schockgefroren", sodass deren räumliche Struktur erhalten bleibt. Anschließend werden aus vielen verschiedenen Blickwinkeln zweidimensionale elektronenmikroskopische Bilder des Objekts aufgenommen, aus denen dann ein dreidimensionales "Bild" rekonstruiert wird.
Die Untersuchungen zeigten, dass die Ribosomen im Polysom dicht gepackt vorliegen und sich so anordnen, dass bestimmte Bauteile - die sogenannten kleinen Untereinheiten - sich zum Inneren des Polysoms hin ausrichten und einander zugewandt sind. Dabei nehmen sie bevorzugt zwei Konfigurationen ein: Entweder liegen die Ribsomen gegeneinander versetzt in einer Ebene (die Untereinheiten liegen "Kopf-an-Kopf"), oder sie sind um 180° gegeneinander verdreht (die Untereinheiten liegen "Kopf-an-Ende"). In beiden Fällen liegen der mRNA Aus- bzw. Eingang benachbarter Ribosomen nah zusammen, sodass das Botenmolekül ohne große Umwege von einem Ribosom zum nächsten gelangt. Sind alle Ribosomen eines Polysoms in der "Kopf-an-Kopf"-Konfiguration einander zugewandt, entsteht eine ausgeprägte schraubenartige Struktur. Bei abwechselnder Anordung mit der "Kopf-an-Ende"-Konfiguration ergibt sich eine flächige Polysomenstruktur (siehe Abbildung).
Das Ablesen der mRNA, die wichtige Übertragung der genetischen Information in die Struktur der Proteine, erfolgt geschützt im Innenraum des Polysoms während sowohl die "Eingänge" durch die die Aminosäuren zu dem Ort in den Ribosomen gelangen, an dem sie verknüpft werden, als auch die "Ausgänge" für die fertigen Proteine dem Zellraum zugewandt und so leicht erreichbar sind.
"Wir haben mit unserer Strukturaufklärung gezeigt, dass die Anordnung der Polysomen dafür sorgt, dass die in benachbarten Ribosomen erzeugten Proteine den größtmöglichen Abstand voneinander einhalten. So wird verhindert, dass sich neu entstehende Proteine miteinander "Verheddern" und Verklumpen", erklärt Hartl. Die bisherige Annahme, hauptsächlich bestimmte Proteine - sogenannte Chaperone - wären dafür zuständig, erscheint vor dem Hintergrund dieser Ergebnisse in einem neuen Licht: Möglicherweise dienen sie weniger dazu, ein Zusammenklumpen im Polysom gebildeter Proteine zu verhindern - hiervor schützt ja schon der strukturell bedingte Abstand zwischen den Proteinsträngen - als vielmehr dem Zweck, fehlerhafte Faltungen innerhalb eines Proteins zu vermeiden und ein Zusammenklumpen mit Proteinen die in anderen Polysomen gebildet wurden, zu verhindern - ein Fehler der in der vollgestopften zellulären Umgebung, wo Polysomen dicht an dicht auftreten, leicht passieren könnte.
Originalveröffentlichung: Florian Brandt et al.; "The Native 3D Organization of Bacterial Polysomes"; Cell 23.1.2009