Neues Nachweisverfahren für Antibiotika-Resistenz: Mutationen geben Leuchtsignale
Anzeigen
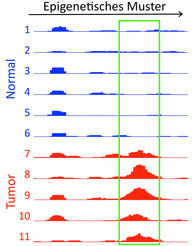
Der Austausch eines einzigen Gen-Bausteins im Erbgut des Tuberkulose-Erregers verursacht Resistenz gegen das Antibiotikum Rifampicin. Wissenschaftler aus dem Deutschen Krebsforschungszentrum und den Universitäten Heidelberg und Bielefeld entwickelten ein hochempfindliches Testverfahren, das diese Genveränderung auf der Ebene des Einzelmoleküls nachweist und damit Auskunft über den Resistenzstatus eines Infizierten gibt.
Wissenschaftler um Dr. Jens-Peter Knemeyer aus der Abteilung Funktionelle Genomanalyse im Deutschen Krebsforschungszentrum kombinieren ein Hybridisierungsverfahren, bei dem sich kleine DNA-Sonden hochspezifisch ausschließlich an die mutierte Gensequenz binden, mit der Technik der konfokalen Mikroskopie: Die DNA-Sonden sind mit einem Fluoreszenzfarbstoff gekoppelt, der unter Laserlicht aufleuchtet. Dieses Leuchtsignal erscheint jedoch nur dann, wenn die Sonde sich an ihre Zielsequenz auf dem Bakterienerbgut anlagert. "Ungebundene" Sondenmoleküle geben kein Signal ab. Jeder dieser Mini-Lichtblitze, die entstehen, wenn sich Sonde und Zielmolekül aneinander binden, weist ein einziges mutiertes DNA-Molekül nach.
Durch die Messung der Dauer und der Abklingzeiten der Lichtblitze unterscheiden die Forscher ein echtes Messergebnis vom allgegenwärtigen Hintergrundleuchten: Aufgrund chemischer Eigenschaften der beteiligten Moleküle klingt die spontane Fluoreszenz wesentlich schneller ab als das Signal der Farbstoff gekoppelten Sonde.
Der Nachweis der Resistenz verursachenden Punktmutation im Erbgut des Tuberkelbazillus ist nur eine von zahlreichen Einsatzmöglichkeiten der so genannten Einzelmolekül-Fluoreszenz-Spektroskopie. Das Verfahren bietet einen großen Vorteil: Statt, wie beim Nachweis der Antibiotika-Resistenz, die Lichtblitze in einer Probenlösung zu erfassen, kann die Untersuchungsmethode auch in lebenden Zellen durchgeführt werden.
Originalveröffentlichung: Nicole Marmé, Achim Friedrich, Matthias Müller, Oliver Nolte, Jürgen Wolfrum, Jörg D. Hoheisel, Markus Sauer, Jens-Peter Knemeyer; "Identification of single-point mutations in mycobacterial 16S rRNA sequences by confocal single-molecule fluorescence spectroscopy."; Nucleic Acids Research Band 34 2006
Weitere News aus dem Ressort Wissenschaft
Meistgelesene News
Weitere News von unseren anderen Portalen
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Spektroskopie
Durch die Untersuchung mit Spektroskopie ermöglicht uns einzigartige Einblicke in die Zusammensetzung und Struktur von Materialien. Von der UV-Vis-Spektroskopie über die Infrarot- und Raman-Spektroskopie bis hin zur Fluoreszenz- und Atomabsorptionsspektroskopie - die Spektroskopie bietet uns ein breites Spektrum an analytischen Techniken, um Substanzen präzise zu charakterisieren. Tauchen Sie ein in die faszinierende Welt der Spektroskopie!

Themenwelt Spektroskopie
Durch die Untersuchung mit Spektroskopie ermöglicht uns einzigartige Einblicke in die Zusammensetzung und Struktur von Materialien. Von der UV-Vis-Spektroskopie über die Infrarot- und Raman-Spektroskopie bis hin zur Fluoreszenz- und Atomabsorptionsspektroskopie - die Spektroskopie bietet uns ein breites Spektrum an analytischen Techniken, um Substanzen präzise zu charakterisieren. Tauchen Sie ein in die faszinierende Welt der Spektroskopie!