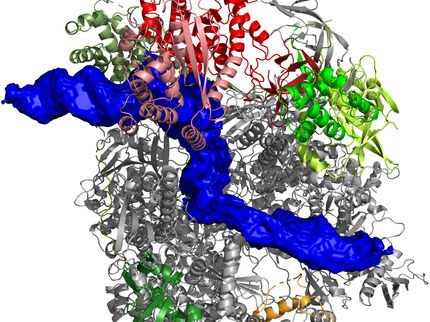
Unbekannte Proteinkomplexe leichter entschlüsseln
Neue Plattform zur Aufklärung von großen Protein- und Nukleinsäurestrukturen bei Infektionen
Anzeigen
Um neue Arzneimittel gegen Infektionskrankheiten entwickeln zu können, müssen Forscher die molekularen Grundlagen verstehen. Wie läuft die Vermehrung eines Erregers ab, welche Wechselwirkungen finden zwischen ihm und der Wirtszelle statt und wie werden diese Vorgänge reguliert? Dazu werden RNA-Proteinkomplexe untersucht. Sie spielen zu verschiedenen Zeitpunkten im Lebenszyklus des Erregers oder der Wirtszelle eine wichtige Rolle und sind so an Infektionsprozessen beteiligt. Forscher des Helmholtz-Zentrums für Infektionsforschung (HZI), der Leibniz Universität Hannover, des EMBL Heidelberg und des Bijvoet Center for Biomolecular Research der Universität Utrecht entwickelten jetzt eine integrierte Strukturanalyse-Plattform, die große Proteinkomplexe auf Basis vielfältiger experimenteller Daten sehr einfach und effektiv berechnen kann. Das sogenannte „M3 Framework“ steht kostenlos für Forscher aus akademischen Instituten und der Industrie zur Verfügung.
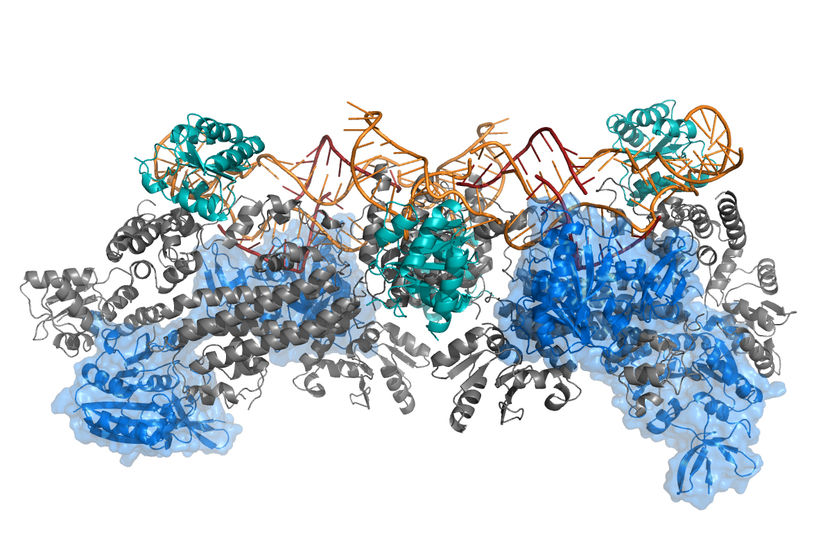
Beispiel für eine komplexe Proteinstruktur, die mit dem M3 Framework berechnet wur das Box C/D-Enzym zur RNA-Methylierung.
HZI/Karaca et al. 2017
Wie winzige Maschinen verrichten Proteine in unserem Körper harte Arbeit. Praktisch alle Prozesse werden durch diese hochspezialisierten Eiweißmoleküle ausgeführt oder gesteuert. Sie übertragen Signale, wandeln Energie um, bringen chemische Reaktionen in Gang oder sorgen für Wachstum und Bewegung. Diese teilweise sehr komplexen Proteinmaschinen, wie beispielsweise RNA-Polymerasen, sind in ihrer Struktur und Funktion nicht einfach zu entschlüsseln. Üblicherweise nutzen Wissenschaftler dafür Methoden der Proteinkristallographie oder der Elektronenmikroskopie. Diese Methoden haben allerdings den Nachteil, dass sie die natürliche Form und Funktion der Proteine und Nukleinsäuren beeinträchtigen können.
Prof. Teresa Carlomagno, Leiterin der Arbeitsgruppe „NMR-basierte Strukturbiologie“ am HZI, verfolgt deshalb eine andere Herangehensweise: Sie erforscht große RNA-Proteinkomplexe in Lösung mit der Kernspinresonanzspektroskopie (kurz NMR für nuclear magnetic resonance-Spektroskopie). Dies ist ein Verfahren zur Untersuchung der elektronischen Umgebung einzelner Atome und der Wechselwirkungen mit den Nachbaratomen. „Der große Vorteil der Methode besteht darin, dass wir komplexe Proteinmaschinen als aktive Enzyme bei der Arbeit mit ihrer natürlichen dynamischen Faltung und Form erleben können“, sagt Teresa Carlomagno.
Das Team um Carlomagno ging noch einen Schritt weiter und entwickelte jetzt eine moderne Plattform, die in der Lage ist, verschiedenartige experimentelle Daten zu integrieren. „Die Daten können beispielsweise aus Mutationsanalysen, der NMR-Spektroskopie, der Elektronenmikroskopie, der Fluoreszenzspektroskopie oder dem Modelling stammen“, sagt Teresa Carlomagno. „Durch die Kombination aller Methoden ist eine Berechnung des Proteinkomplexes möglich, der uns eine erste Idee gibt, womit wir es zu tun haben. Gleichzeitig gibt die M3-Plattform den Wissenschaftlern auch Hinweise, wenn Daten noch nicht ausreichen und weitere Experimente notwendig sind, um die Proteinmaschine akkurat zu beschreiben.“
Viel Gelegenheit zur Forschung und zum Einsatz der M3-Plattform haben Teresa Carlomagno und ihr Team derzeit in engen Kooperationen mit weiteren HZI-Arbeitsgruppen. Gemeinsam mit dem Team von Prof. Rolf Müller am Helmholtz-Institut für Pharmazeutische Forschung Saarland in Saarbrücken untersuchen die Forscher beispielsweise bislang unbekannte Proteinmaschinen, die antiinfektive Wirkstoffe bilden. Diese werden dringend in der Entwicklung neuer Antibiotika benötigt.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft
Meistgelesene News
Weitere News von unseren anderen Portalen
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Spektroskopie
Durch die Untersuchung mit Spektroskopie ermöglicht uns einzigartige Einblicke in die Zusammensetzung und Struktur von Materialien. Von der UV-Vis-Spektroskopie über die Infrarot- und Raman-Spektroskopie bis hin zur Fluoreszenz- und Atomabsorptionsspektroskopie - die Spektroskopie bietet uns ein breites Spektrum an analytischen Techniken, um Substanzen präzise zu charakterisieren. Tauchen Sie ein in die faszinierende Welt der Spektroskopie!

Themenwelt Spektroskopie
Durch die Untersuchung mit Spektroskopie ermöglicht uns einzigartige Einblicke in die Zusammensetzung und Struktur von Materialien. Von der UV-Vis-Spektroskopie über die Infrarot- und Raman-Spektroskopie bis hin zur Fluoreszenz- und Atomabsorptionsspektroskopie - die Spektroskopie bietet uns ein breites Spektrum an analytischen Techniken, um Substanzen präzise zu charakterisieren. Tauchen Sie ein in die faszinierende Welt der Spektroskopie!