Was passiert in der lebenden Zelle?
Mit „Molecular Activity Painting” Zellbewegungen durch einen Lichtpuls-Schalter steuern und beobachten
Die Plasmamembran ist eine Schaltstelle für Signalkaskaden, die wichtige Zellprozesse kontrollieren. Sie ist allerdings ein sehr fluides Medium, was die Erforschung solcher Vorgänge schwierig macht. Deutsche Wissenschaftler haben jetzt eine molekulare „Malpinsel”-Technik entwickelt, mit der sie Signalkaskaden steuern und beobachten können. Wie sie in der Zeitschrift Angewandte Chemie berichten, konnten sie mit ihrem modularen System aus membranverankerten, lichtaktivierbaren molekularen Bausteinen Kontraktionsmuster in lebenden Zellen gezielt auslösen und beobachten.
© Wiley-VCH
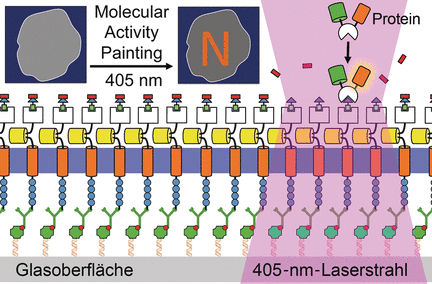
Die Plasmamembran umschließt als Lipidbarriere jede einzelne Zelle. Membranproteine regeln und kontrollieren den Durchlass von Wasser, Ionen, Proteinen und anderen Komponenten. Signale aus der äußeren Umgebung werden durch membranständige Rezeptoren ins Zellinnere übertragen, um zum Beispiel die Zellbewegung oder Differenzierung von Zellen zu steuern. Solche Signalkaskaden lassen sich auf molekularer Ebene nur sehr schwer beobachten, weil die Proteine innerhalb der fluiden Lipidschicht der Plasmamembran sehr schnell wandern können. Die Teams von Leif Dehmelt am Max-Planck-Institut für molekulare Physiologie in Dortmund und Yao-Wen Wu am Chemical Genomics Centre der Max-Planck-Gesellschaft gehen daher einen neuen Weg: Sie versehen einen am Zellsubstrat verankerten künstlichen Rezeptor mit einem speziell entwickelten, modular aufgebauten Molekülsystem. Ein Lichtpuls aktiviert die Bausteine, die nun im Zellinneren eine lokalisierte Signalkaskade bis hin zur Bewegung von Bestandteilen des Zytoskeletts auslösen - von außen sichtbar als „molekularer Pinselstrich” auf der Membran. Diese Technolgie nennen sie „Molecular Activity Painting” oder kurz: MAP.
Das Herzstück von MAP ist ein lösliches, aus vier Teilen aufgebautes Molekül. Es enthält eine Chloralkan-Kohlenwasserstoffkette, eine Polymerbrücke (PEG), eine Molekülgruppe namens Trimethroprim (TMP) und eine lichtempfindliche Gruppe namens Nvoc. Dieses Multifunktionsmolekül erfüllt mehrere Aufgaben: Durch seine Chloralkylgruppe bindet es an einen künstlichen, fest am Zellsubstrat verankerten Rezeptor. Ein einziger Lichtpuls entfernt die Nvoc-Gruppe, und die dadurch freigesetzte TMP-Einheit rekrutiert ein zytosolisches Konstrukt, das wiederum eine Signalkaskade in der Zelle auslöst. Das ganze System hat nur einen Zweck: Steuerung und Visualisierung von molekularen Vorgängen in der lebenden Zelle.
Die Wissenschaftler konnten durch MAP die lokalisierte Kontraktion von Actomyosin in Säugerzellen steuern. Dabei „malten” sie den Buchstaben N auf die Plasmamembran. „‚Molecular Activity Painting’ ermöglicht schaltbare, strukturierte Störungen von regulatorischen Netzwerken im Mikrometer-Maßstab”, erklären die Autoren.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.