Auf der molekularen Streckbank
Anzeigen
Das Erbgutmolekül enthält den Bauplan des Lebens. Wie der Bauplan in der Zelle verpackt ist, bestimmt auch, welche Gene aktiv sind und welche stumm geschaltet werden. Gerät die Struktur durcheinander, können Krankheiten wie etwa Krebs entstehen. Münchner Wissenschaftlerinnen und Wissenschaftlern ist es gelungen, mithilfe einer aus DNA konstruierten molekularen Pinzette Interaktionen auf der ersten Verpackungsebene des Erbguts zu messen.
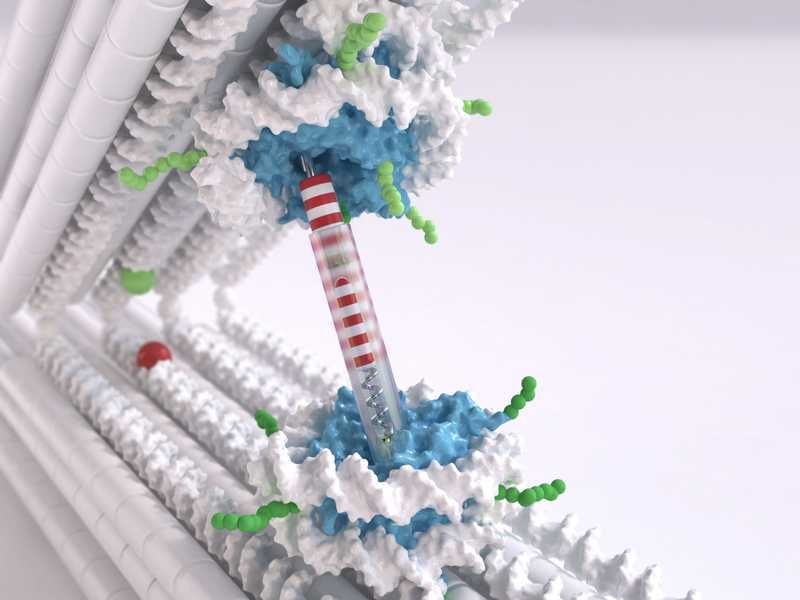
Die Pinzetten-Struktur besteht aus zwei starren DNA-Balken, die durch ein Gelenk verbunden sind.
Chris Hohmann, NIM / Dietz Lab, TUM
Zwei Meter des Moleküls Desoxyribonukleinsäure (DNA), der Trägerin unserer Erbinformation, befinden sich in jedem Kern einer menschlichen Zelle. Die DNA muss daher sorgsam verpackt werden. Sie wickelt sich dabei zunächst um bestimmte Proteine. Diese mit kleinen Spulen vergleichbaren Strukturen aus DNA und Proteinen werden als Nukleosomen bezeichnet. Die Nukleosomen sind miteinander durch Abschnitte nicht aufgewickelter DNA verbunden – unter dem Elektronenmikroskop gleicht die in Nukleosomen verpackte DNA einer Perlenkette.
Wie die Nukleosomen miteinander in Wechselwirkung stehen und welche übergeordneten Strukturen sich daraus ergeben, ist noch nicht vollständig geklärt. Ein Team von Wissenschaftlerinnen und Wissenschaftlern um Hendrik Dietz von der Technischen Universität München (TUM) und Philipp Korber von der Ludwig-Maximilians-Universität München (LMU) ist es nun gelungen, einen Beitrag zur Lösung dieses Rätsels zu leisten: Zum ersten Mal konnten sie die Anziehungskräfte direkt messen, die zwischen den Nukleosomen herrschen.
Nukleosomen werden in die Pinzette eingehängt
Dietz, Inhaber des Lehrstuhls Experimentelle Biophysik an der TUM, nutzt DNA als Baumaterial und konstruiert damit molekulare Strukturen; diese Technik ist unter dem Begriff DNA-Origami bekannt. Um nun die Wechselwirkungen zwischen den Nukleosomen messen zu können, entwickelten er und sein Team eine Pinzetten-Struktur, die aus zwei starren DNA-Balken besteht, die durch ein Gelenk verbunden sind. Pro Balken wurde jeweils eine Nukleosomenstruktur eingehängt. "Wir können die Position und Orientierung der Nukleosomen in den DNA-Pinzetten sehr genau einstellen", sagt Dietz. "Das ist sehr wichtig, um die Interaktionen wirklich messen zu können."
Nukleosomenstrukturen zu entwickeln, die sich in die Pinzette einhängen lassen, war die Herausforderung, der sich die Forscher an der LMU stellten. "Normalerweise gibt es beim Nukleosom zwei recht nah beieinanderliegende Enden des aufgerollten DNA-Doppelstrangs", erklärt Korber. "Aber was wir brauchten, waren zwei herausstehende Einzelstränge mehr in der Mitte. Das war nicht trivial, da dies die Struktur destabilisieren kann. Corinna Lieleg aus unserem Team ist es aber gelungen, die richtigen Stellen für diese Griffe zu finden."
Die Forscher konnten so eine sehr schwache Interaktion der Nukleosomen messen, die bei 1,6 kcal/mol mit einer Reichweite von etwa 6 Nanometer liegt. Die Orientierungen der Nukleosomen zueinander hatten kaum Einfluss auf die Interaktion. Allerdings schwächten bestimmte chemische Veränderungen der Nukleosomen die Wechselwirkungen weiter.
Gibt es die 30-Nanometer-Faser wirklich?
Das Ergebnis könnte dazu beitragen, einen aktuellen Disput in der Wissenschaft zu klären. Die bisher gängige Theorie besagt, dass die Nukleosomen gemeinsam mit weiteren Proteinen eine Art Superspirale mit einem Durchmesser von 30 Nanometern bilden, die sogenannte 30-Nanometer-Faser. Diese nächsthöhere Strukturebene konnte aber noch nie in der lebenden Zelle beobachtet werden. Ob die DNA-Verpackung, das Chromatin, dort wirklich eine solche Superspirale annimmt, ist momentan sehr umstritten. Die geringen Kräfte zwischen den Nukleosomen, die die Forscher nun gemessen haben, sprechen eher gegen die gängige Theorie. "Unsere Daten deuten auf sehr weiche, leicht durch äußere Einflüsse deformierbare Strukturen", sagt Dietz. "Wir können die aktuelle Diskussion mit unserer Arbeit zwar nicht abschließend klären, aber doch wichtige Hinweise hinzufügen und auch ein paar Modelle ausschließen."
Die Frage, wie die Überstruktur der Nukleosomen aussieht, ist von fundamentaler Bedeutung. Nur die Gene, die in einer relativ wenig kompakten Chromatinstruktur liegen, sind "aktiv", was bedeutet, dass die dort codierten Proteine wirklich in der zelleigenen Fabrik produziert werden.
In Krebszellen gerät der Ableseprozess durcheinander
"In den vergangenen zehn Jahren ist immer deutlicher geworden, dass viele Veränderungen und Mutationen, die dazu führen, dass Zellen zu Krebszellen werden, auf dieser Ebene stattfinden", sagt Korber. In einer Krebszelle geraten die zellulären Entscheidungen, welche Gene aktiv und welche inaktiv sind, durcheinander. Abschnitte, die nicht zugänglich sein sollten, liegen frei und umgekehrt. "Wenn aber nur die Verpackung und nicht die Gene selbst fehlerhaft ist, gibt es die therapeutische Hoffnung, dass man die Verpackung wieder ändern kann." Eine Heilung wäre sehr viel schwieriger, wenn die Gene selbst vollständig aus dem Genom gelöscht wären.
Die Forscher wollen die molekularen Pinzetten, die sie für Messungen der Kräfte zwischen Nukleosomen verwendet haben, auch zur Untersuchung anderer Strukturen einsetzen. "In der Biologie ist es immer wichtig, welche Orientierung Strukturen zueinander haben", sagt Korber. "Jetzt haben wir eine Art molekulare Streckbank, mit der wir gezielt die Orientierung der räumlichen Anordnung zueinander kontrollieren können."
In einem weiteren Versuch haben die Forscher auch die Kraft gemessen, die für das Abrollen der DNA aus dem Nukleosom nötig ist. Die Forscher konnten so zeigen, dass es mithilfe des Messsystems möglich ist, sowohl Kräfte zwischen Molekülen als auch innerhalb der Moleküle zu messen.
Originalveröffentlichung
Jonas J. Funke, Philip Ketterer, Corinna Lieleg, Philipp Korber, and Hendrik Dietz; "Exploring Nucleosome Unwrapping Using DNA Origami"; Nano Letters; 2016
Funke, Jonas J. and Ketterer, Philip and Lieleg, Corinna and Schunter, Sarah and Korber, Philipp and Dietz, Hendrik; "Uncovering the forces between nucleosomes using DNA origami"; Science Advances; 2016