Computersimulation macht kurzlebige Proteinstrukturen sichtbar
Chemikern der Universität Basel ist es gelungen, mit Computersimulationen kurzlebige Strukturen in Eiweissmolekülen aufzuklären. Die Forscher berichten, wie mit Computersimulationen atomare Details der Wirkungsweise von Proteinen verstanden werden können.
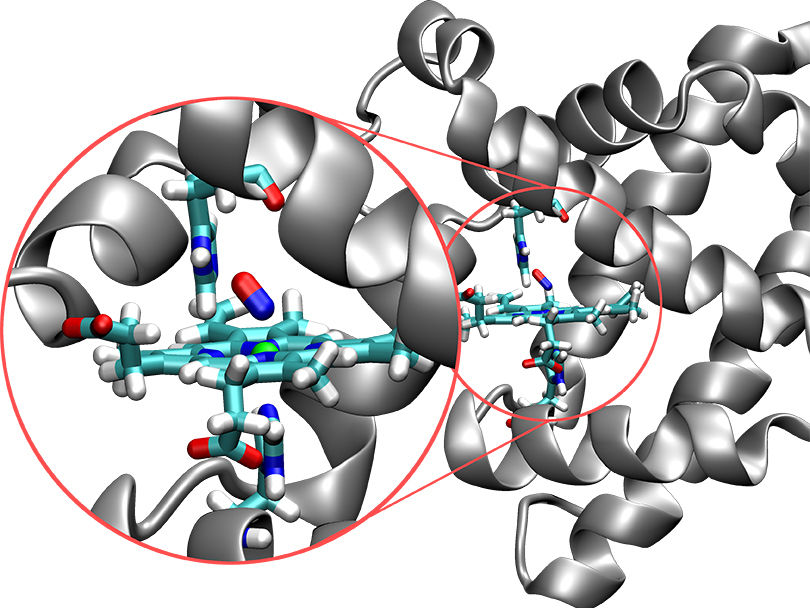
Die Struktur des Proteins Myoglobin (silber) mit dem eingebetteten aktiven Zentrum (farbig). Das Sickstoffmolekül (rot/blau) ist in dieser Abbildung an das Eisenatom (grüne Kugel) gebunden.
Universität Basel, Departement Chemie
Mithilfe der computergestützten Chemie ist es möglich, die Bewegung einzelner Atome einer Verbindung zu charakterisieren. Modernste Simulationstechniken erlauben es heute, die Dynamik von Molekülen und Systemen mit hunderttausenden von Atomen quantitativ zu beschreiben. Diese Techniken sind vor allem bei denjenigen Zuständen wichtig, die wegen ihrer kurzen Lebensdauer experimentell nur schwer direkt beobachtbar sind. Hier liefern Computersimulationen wertvolle, weiterführende Einsichten.
Die Funktion eines Eiweissmoleküls (Proteins) wird durch dessen Struktur und Dynamik bestimmt. Dabei ist von besonderer Bedeutung, dass man die für die Wirkungsweise relevanten Strukturen und molekularen Vorgänge im sogenannten aktiven Zentrum kennt – also an der Stelle, an der chemische Reaktionen stattfinden. Das Bilden und Brechen chemischer Bindungen ist ein dynamischer Prozess und hat Strukturänderungen zur Folge. Die beobachtbare Dynamik endet in der Regel in stabilen (energiearmen) Zuständen, welche durch einen oder mehrere metastabile (energiereichere) Zwischenschritte erreicht werden. Ob sich ein metastabiler Zustand direkt experimentell nachweisen lässt oder nicht, hängt von dessen Lebensdauer ab. Ist diese zu kurz, stehen nur indirekte Nachweismethoden zur Verfügung.
Computer berechnet atomare Geometrie
Ein Forscherteam um Prof. Markus Meuwly vom Departement Chemie der Universität Basel hat nun mit sogenannt reaktiven Molekulardynamik-Simulationen das räumliche und zeitliche Verhalten des Proteins Myoglobin charakterisiert. Myoglobin ist für den Sauerstofftransport innerhalb der Zellen wichtig und kommt hauptsächlich im Muskelgewebe vor. Stickstoffmonoxid, das in den Zellen gebildet wird, ist ein kurzlebiger und reaktiver Botenstoff, der unter anderem eine wichtige Rolle bei der Erweiterung der Blutgefässe unter Sauerstoffmangel spielt.
«Zwar ist der Bindungsprozess von Stickstoffmonoxid an Myoglobin bereits gut experimentell untersucht, was für das Eichen von Computersimulationen wichtig ist», erklärt Prof. Meuwly. «Auch die Existenz von metastabilen Zwischenstufen konnte experimentell nachgewiesen werden. Aber nur mithilfe unserer Simulationen lassen sich die Strukturen solcher Zwischenstufen, ihre Dynamik und damit die Funktion des Proteins weitergehend entschlüsseln.»
Somit bilden Computersimulationen zusammen mit experimentellen Beobachtungen die Grundlage für das Verständnis komplexer chemischer und biologischer Systeme. Solch kombinierte Ansätze sind denn auch Ausgangspunkt für die Behandlung weitergehender Fragen, beispielsweise zur Anpassung und Optimierung von Proteinen oder pharmazeutischen Wirkstoffen. Dies setzt ein Verständnis der zugrunde liegenden Prozesse auf molekularer und atomarer Ebene voraus.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft
Diese Produkte könnten Sie interessieren

Antibody Stabilizer von CANDOR Bioscience
Protein- und Antikörperstabilisierung leicht gemacht
Langzeitlagerung ohne Einfrieren – Einfache Anwendung, zuverlässiger Schutz

DynaPro NanoStar II von Wyatt Technology
NanoStar II: DLS und SLS mit Touch-Bedienung
Größe, Partikelkonzentration und mehr für Proteine, Viren und andere Biomoleküle

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.
Meistgelesene News
Weitere News von unseren anderen Portalen
Zuletzt betrachtete Inhalte

Patentstreit um mRNA-Technologie - Curevac und Biontech streiten nun auch vor Bundespatentgericht
Myostatin
























































