3D-Struktur von Biomolekülen vorhersagen
KIT-Forscher erhalten „Google Faculty Research Award“ für Forschung zur Raumstruktur von Biomolekülen
Ohne Biomoleküle kein Leben: In jeder Zelle eines Organismus arbeitet eine Heerschar winziger Maschinen, die etwa Sauerstoff transportieren, Nährstoffe verwerten oder Schäden in der Erbinformation reparieren. Entscheidend für die Funktion des molekularen Werkzeugkastens der Zelle ist die räumliche Struktur der Mini-Maschinen. Forscher des Karlsruher Instituts für Technologie (KIT) haben eine effektive Methode entwickelt, mit der sie durch Analyse einfach zu gewinnender experimenteller Daten die 3-dimensionale Struktur von Biomolekülen vorhersagen können.
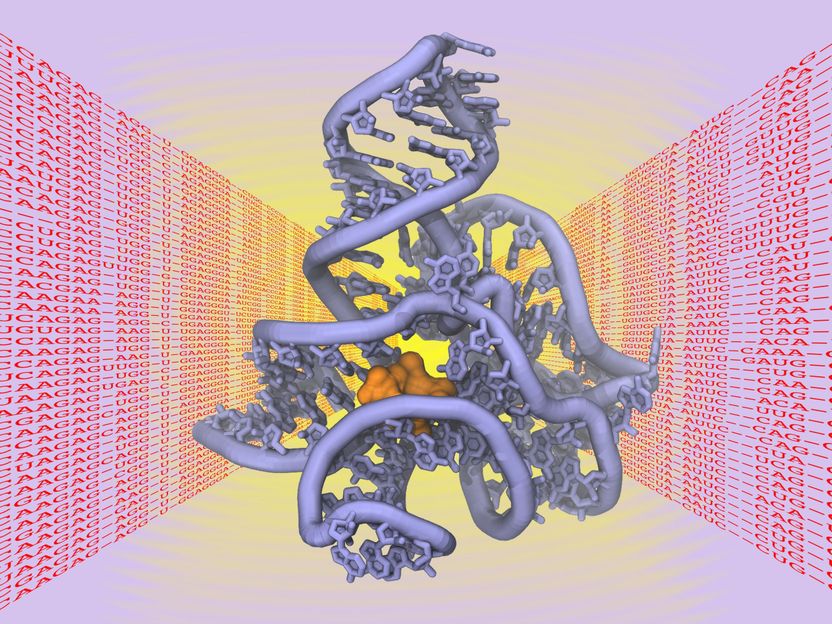
Die räumliche Struktur von Biomolekülen wie Proteinen, DNS oder RNS ist entscheidend für ihre Funktion als „molekulare Maschinen“ in der Zelle.
Alexander Schug
Wie funktioniert Leben auf der molekularen Ebene? Um diese Frage beantworten zu können, die für die Biologie, Pharmazie und Medizin von zentraler Bedeutung ist, muss man in einem ersten Schritt die dreidimensionale Struktur von Biomolekülen kennen. Denn Biomoleküle wie Proteine, DNS oder RNS sind die molekularen Maschinen in Zellen, die diverse Aufgaben wie Sauerstofftransport, Muskelaktivität oder das Speichern und Ausführen genetischer Information erfüllen.
Entscheidend für die Funktion dieser mikroskopisch kleinen Maschinen ist ihre räumliche Struktur. So binden etwa bestimmte Proteine ihre „Zielsubstanz“ nur deshalb so hochspezifisch, weil diese genau – wie ein Schlüssel zum Schloss – in die aktive Bindungsregion des Proteins passt.
Die experimentellen Methoden zur Bestimmung der räumlichen Struktur von Biomolekülen sind mittlerweile zwar sehr ausgefeilt, unterliegen aber dennoch technischen Beschränkungen.
Die Forschungsgruppe um Alexander Schug vom Steinbuch Centre of Computing (SCC) am KIT hat einen alternativen Ansatz entwickelt, der auf statistischen Analysen großer Datenmengen von Biomolekülen aus verschiedenen Organismen beruht, die experimentell sehr leicht zu gewinnen sind. Algorithmen analysieren diese Daten auf Mutationsmuster, die eine Vorhersage der räumlichen Struktur ermöglichen. Diese Arbeiten wurden nun mit dem Forschungspreis von Google ausgezeichnet.
„Unsere Arbeit ist ein schönes Beispiel für die Interdisziplinarität in moderner Forschung: Wir haben Methoden aus der theoretischen Physik und Informatik auf eine Fragestellung der molekularen Lebenswissenschaften übertragen“, sagt Alexander Schug. „Wir erhoffen, dass unsere detaillierten Strukturvorhersagen aufgrund der Bedeutung von Biomolekülen für viele Krankheiten neben der Relevanz für die Grundlagenforschung auch in der pharmakologischen und medizinischen Forschung Anwendung finden werden.“
Der „Google Faculty Research Award“ wird weltweit an ausgewählte universitäre Forschungsprojekte aus der Informatik, den Ingenieurwissenschaften und verwandten Bereichen vergeben und soll die universitäre Forschung sowie den Austausch mit den Universitäten unterstützen. Mit dem Preis fördert Google das Forschungsprojekt der Gruppe um Alexander Schug über ein Jahr und stellt dafür rund 50.000 US-Dollar zur Verfügung.
Meistgelesene News
Weitere News aus dem Ressort Wissenschaft

Holen Sie sich die Life-Science-Branche in Ihren Posteingang
Mit dem Absenden des Formulars willigen Sie ein, dass Ihnen die LUMITOS AG den oder die oben ausgewählten Newsletter per E-Mail zusendet. Ihre Daten werden nicht an Dritte weitergegeben. Die Speicherung und Verarbeitung Ihrer Daten durch die LUMITOS AG erfolgt auf Basis unserer Datenschutzerklärung. LUMITOS darf Sie zum Zwecke der Werbung oder der Markt- und Meinungsforschung per E-Mail kontaktieren. Ihre Einwilligung können Sie jederzeit ohne Angabe von Gründen gegenüber der LUMITOS AG, Ernst-Augustin-Str. 2, 12489 Berlin oder per E-Mail unter widerruf@lumitos.com mit Wirkung für die Zukunft widerrufen. Zudem ist in jeder E-Mail ein Link zur Abbestellung des entsprechenden Newsletters enthalten.






















































