3-D-Nanostruktur eines Knochens sichtbar gemacht
Knochen bestehen aus winzigen Fasern, die etwa tausend Mal feiner sind als ein menschliches Haar. Mit einer neuartigen computerbasierten Auswertungsmethode konnten Forschende des Paul Scherrer Instituts PSI zum ersten Mal die Ordnung und Ausrichtung dieser Nanostrukturen innerhalb eines gesamten Knochenstücks sichtbar machen.
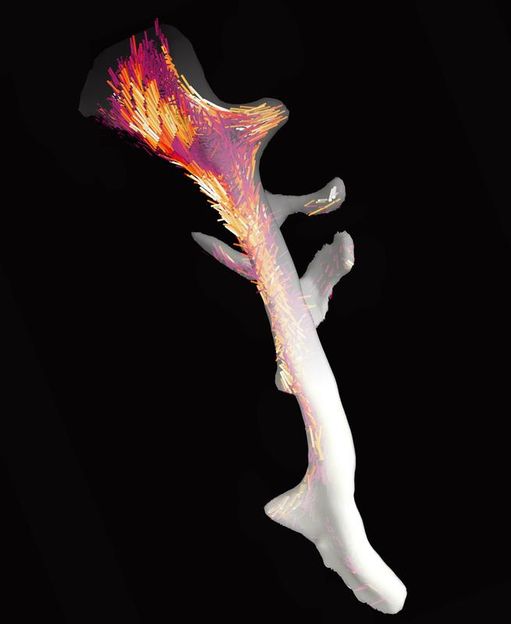
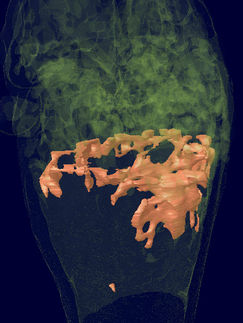
Der Knochen und seine Nanostruktur: Dank ihrer neu entwickelten Auswertungsmethode konnten Forschende am PSI die Ausrichtung der winzigen Kollagenfibrillen kartieren.
Paul Scherrer Institut/Marianne Liebi
Die Anordnung der Nanostruktur eines dreidimensionalen Objektes lässt sich dank einer neuen Methode von Forschern am Paul Scherrer Institut PSI sichtbar machen. An einem rund zweieinhalb Millimeter langen Stück eines menschlichen Rückenwirbels haben die Forscher dies in Zusammenarbeit mit Knochen-Biomechanikern der ETH Zürich und der Universität Southampton, England, demonstriert. Knochen bestehen aus winzigen Fasern, sogenannten Kollagenfibrillen. Deren dreidimensionale lokale Ausrichtung wurde nun entlang des kompletten Knochenstücks sichtbar gemacht. Diese Eigenschaften bestimmen massgeblich die Stabilität eines Knochens. Somit könnte die Abbildungstechnik unter anderem der Erforschung der Osteoporose zugutekommen. Allgemein eignet sie sich nicht nur zur Untersuchung biologischer Objekte, sondern auch zur Entwicklung zukunftsträchtiger Materialien.
Die Daten wurden an der Synchrotron Lichtquelle Schweiz SLS des PSI gewonnen, wo das Knochenstück mit einem extrem feinen und intensiven Röntgenstrahl durchleuchtet wurde. Dieser Strahl rastert über die Probe und vermisst sie so Punkt für Punkt. Dadurch kann an jedem Messpunkt die lokale Nanostruktur bestimmt werden.
Der entscheidende Schritt von 2-D zu 3-D
Bisher liessen sich nur zweidimensionale Proben auf diese Art abrastern und untersuchen. Klassischerweise werden die Untersuchungsobjekte daher in sehr dünne Scheiben geschnitten. "Allerdings lässt sich nicht jedes Objekt beliebig dünn schneiden", erklärt Projektbetreuer Manuel Guizar-Sicairos. "Und manchmal zerstört oder verändert man dabei gerade die Nanostruktur, die man untersuchen wollte." Auch ist ganz grundsätzlich eine zerstörungsfreie Methode vorzuziehen, bei der also das Untersuchungsobjekt nach der Messung noch zur Verfügung steht.
Um nun auch dreidimensionale Objekte abbilden zu können, rasterten die PSI-Forschenden ihre Probe immer wieder, drehten sie jedoch zwischen zwei Aufnahmen jeweils um einen kleinen Winkel. So erhielten sie Messdaten aus allen Raumrichtungen, die ihnen erlaubten, das dreidimensionale Objekt inklusive seiner Nanostruktur nachträglich im Computer zu rekonstruieren.
Damit knüpft die neue Messmethode der PSI-Forschenden an ein Prinzip aus der Computertomographie (CT) an. Auch dort werden zunächst viele Röntgenaufnahmen eines Patienten oder Objekts aus verschiedenen Richtungen angefertigt und anschliessend per Computerauswertung zu den gewünschten Bildern zusammengesetzt. Allerdings nutzt die herkömmliche Computertomographie keinen feinen Röntgenstrahl, sondern das Objekt wird flächig beleuchtet.
Dadurch lässt sich per Computertomographie zwar die variierende Dichte des Materials abbilden, nicht jedoch die Ausrichtung der zugrunde liegenden Nanostruktur. Letzteres wird erst möglich durch den schmalen, intensiven Röntgenstrahl der SLS sowie durch hochmoderne Detektoren.
Bilder entstehen dank mathematischer Algorithmen
Der komplexeste Schritt war jedoch, aus der immensen Zahl der Daten per Computer ein Bild der dreidimensionalen Probe zusammenzusetzen. Hierfür entwickelten die Forschenden einen eigenen aufwendigen mathematischen Algorithmus. "Der Röntgenstrahl durchquert immer die Probe in ihrer ganzen Tiefe und wir sehen nur das Endergebnis", erklärt Marianne Liebi, Erstautorin der Studie. "Wie die dreidimensionale Struktur aussieht, das müssen wir nachträglich herausfinden."
Liebis Algorithmus sucht für jeden Punkt im Inneren der Probe die Struktur, die am besten den gemessenen Daten entspricht. Dabei nutzten die Forschenden den Umstand aus, dass sie von einer gewissen Symmetrie bei der Anordnung der Kollagenfibrillen im Knochen ausgehen konnten und reduzierten dadurch ihre Daten auf ein berechenbares Mass. Dennoch blieben 2,2 Millionen Parameter. Diese wurden per Computerprogramm optimiert, bis die Forschenden das Bild der Probe erhielten, das die Messung am besten erklärte.
"Ich war überrascht, dass nach so viel purer Mathematik ein Bild entstand, das wirklich wie ein Knochen aussah," so Liebi. "Die Details darin sahen direkt einleuchtend aus."
Wie eine Landkarte der Vegetationszonen
Während die klassische Computertomographie Graustufen-Bilder erzeugt, entstehen mit der neuen Methode quasi bunte Abbildungen mit deutlich mehr Information: Die farbigen Linien zeigen die Orientierung auf der Nanoskala an und geben sogar Auskunft über das Ausmass der Orientierung – ob also die Kollagenfibrillen an einem bestimmten Punkt grösstenteils, teilweise oder gar nicht parallel zueinander liegen.
"Wir können zwar nicht jede einzelne Kollagenfibrille direkt abbilden, aber wir brauchen das auch gar nicht", erklärt Guizar-Sicairos. "Unsere Bildgebung gleicht eher einer Landkarte der Vegetationszonen. Auch dafür wird jeweils über ein gewisses Areal gemittelt und gesagt: Hier gibt es vor allem Nadelbäume, dort Laubbäume und dort Mischwälder." So lässt sich die Vegetation ganzer Kontinente kartieren, ohne jede einzelne Baumart bestimmen zu müssen.
Analog lässt sich sagen, dass bei herkömmlichen mikro- und nanoskopischen Methoden diese Abbildung einzelner Bäume noch nötig war. Darum galt: Je kleiner die Struktur eines Objekts, desto kleiner musste auch der Bildausschnitt sein. Durch ihre neue Methode ist es den PSI-Forschenden gelungen, diese Regel zu umgehen: Von dem mit blossem Auge sichtbaren Knochenstück haben sie die Anordnung der Nanostruktur in einem einzigen Bild festgehalten.
Zeitgleich mit ihrer Veröffentlichung erscheint ebenfalls im Fachblatt Nature eine Publikation unter Federführung eines anderen Forscherteams, bei der Liebi und Guizar-Sicairos Mitautoren sind. Hierin wird eine alternative Auswertungsmethode vorgestellt, die zu einem ähnlichen Forschungsergebnis führt: Den Forschenden gelang es, die dreidimensionale innere Nanostruktur eines menschlichen Zahns zu bestimmen.
Originalveröffentlichung
M. Liebi, M. Georgiadis, A. Menzel, P. Schneider, J. Kohlbrecher, O. Bunk and M. Guizar-Sicairos; "Nanostructure surveys on macroscopic specimens by small-angle scattering tensor tomography"; Nature; 2015
F. Schaff, M. Bech, P. Zaslansky, C. Jud, M. Liebi, M. Guizar-Sicairos and F. Pfeiffer; "Six-dimensional real and reciprocal space small-angle X-ray scattering tomography"; Nature; 2015