Forscher röntgen Entzündungshemmer
Studie bestimmt erstmals molekulare Struktur sogenannter Spiegelmere
Mit DESYs Röntgenquelle PETRA III haben Forscher erstmals die räumliche Struktur von zwei medizinisch vielversprechenden Molekülen aus der neuen Wirkstoffgruppe der Spiegelmere entschlüsselt. Die Ergebnisse ermöglichen ein tieferes Verständnis der Funktion dieser Wirkstoffe, die sich bereits als Entzündungshemmer in der klinischen Erprobung befinden. Die Wissenschaftler der Universitäten Hamburg und Aarhus (Dänemark) sowie der Berliner Biotech-Firma NOXXON stellen ihre Analysen im britischen Fachblatt „Nature Communications“ vor.
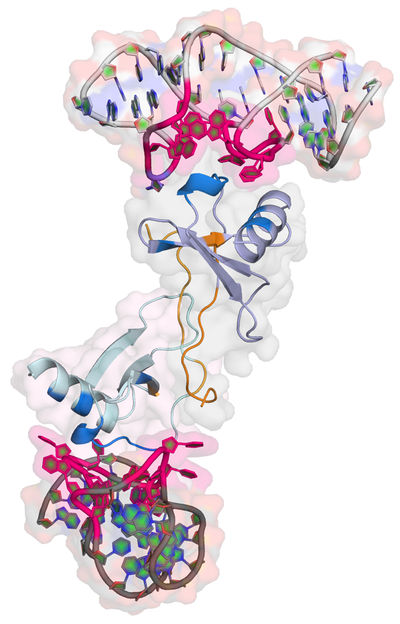
Struktur des Spiegelmers NOX-E36, gebunden an das Entzündungsprotein CCL2.
Dominik Oberthür/CFEL
Spiegelmere sind eine junge Gruppe von Wirkstoffen. Sie beruhen auf Bausteinen der Nukleinsäuren (RNA oder DNA), die im Organismus verschiedene Aufgaben erfüllen, etwa die Speicherung und Übertragung von Erbinformationen oder die Regulierung von Genen. Künstlich hergestellte RNA- oder auch DNA-Moleküle, sogenannte Aptamere, können sehr spezifisch an bestimmte Eiweißmoleküle (Proteine) binden und damit deren Funktion blockieren. Gleichzeitig werden sie vom Organismus toleriert, da sie aus natürlichen Bausteinen aufgebaut sind. Aptamere gelten daher als Erfolg versprechende medizinische Wirkstoffkandidaten. Seit 2006 ist bereits ein Aptamer zur Behandlung der altersbedingten Makula-Degeneration (AMD) zugelassen, einer Augenkrankheit, die zu Blindheit führen kann.
RNA- und DNA-Moleküle werden im Körper in der Regel sehr schnell abgebaut, was den Einsatz als medizinische Wirkstoffe stark einschränkt. Allerdings existieren von den meisten Biomolekülen zwei spiegelbildliche Varianten, eine L-Form und eine D-Form. RNA kommt im Organismus stets nur in der D-Form vor, Proteine dagegen nur in der L-Form. Erzeugt man im Labor Aptamere in der L-Form, werden diese vom Organismus nicht abgebaut. Diese spiegelbildlichen L-Aptamere heißen Spiegelmere. „Ein Vorteil ist, dass Spiegelmere nicht von körpereigenen Enzymen angegriffen werden“, erläutert Prof. Christian Betzel von der Universität Hamburg.
„Spiegelmere werden im Labor über ein ausgeklügeltes evolutives Verfahren identifiziert und optimiert. Allerdings gab bislang keinerlei Strukturinformationen von Spiegelmeren“, betont Erstautor Dr. Dominik Oberthür vom Center for Free-Electron Laser Science CFEL, einer Kooperation von DESY, Max-Planck-Gesellschaft und Universität Hamburg. Wäre die räumliche Struktur eines Spiegelmers zusammen mit der Bindungsstelle an seinem Zielprotein bekannt, ließe sich der genaue Wirkmechanismus aufklären und bei Bedarf die Form des Wirkstoffs gezielt optimieren.
Das Team um Betzel hat mit dem hellen Röntgenlicht von PETRA III nun ein Spiegelmer der Firma NOXXON mit der Bezeichnung NOX-E36 analysiert. Es hemmt ein Protein namens CCL2, das an zahlreichen Entzündungsprozessen im Körper beteiligt ist. „Wenn man so ein Entzündungsprotein gezielt mit einem Spiegelmer blockiert, hat man gute Chancen, die Entzündung im Körper herunterzufahren“, erläutert Betzel. NOX-E36 ist bereits erfolgreich in einer sogenannten Phase-IIa-Studie mit Patienten getestet worden.
Für die Strukturanalyse des neuartigen Wirkstoffs züchteten die Wissenschaftler zunächst Kristalle aus dem Spiegelmer und dem daran gebundenen Protein CCL2. „Die Kristallisation war eine Herausforderung“, berichtet Betzel. Denn die meisten Biomoleküle lassen sich nur sehr widerstrebend in Kristallform zwingen, weil das ihrer natürlichen Funktion widerspricht.
Die Kristalle untersuchte das Team an der Messstation P13 bei DESY, die vom Europäischen Laboratorium für Molekularbiologie EMBL betrieben wird. Wird so ein Kristall mit Röntgenlicht beleuchtet, entsteht ein charakteristisches Beugungsmuster, aus dem sich die Struktur der Biomoleküle errechnen lässt – in diesem Fall also die Form des Spiegelmers, gebunden an sein Zielmolekül. Auf dieselbe Weise analysierte eine Gruppe um Laure Yatime von der Universität Aarhus ein zweites Spiegelmer: NOX-D20 koppelt an das Protein C5a, das ebenfalls an zahlreichen Entzündungsprozessen beteiligt ist.
Die Analysen zeigen die räumliche Struktur der beiden Spiegelmere mit einer Detailgenauigkeit von 0,2 Nanometern (millionstel Millimetern), das ist in der Größenordnung einzelner Atome. „Ich bin begeistert, endlich eine hochauflösende Visualisierung der bemerkenswerten Formen von zwei Spiegelmer-Wirkstoffkandidaten zu haben“, betont der Gründer und wissenschaftliche Leiter von NOXXON, Dr. Sven Klussmann, der bei beiden Fachveröffentlichungen Mitautor ist. „Die Strukturdaten liefern uns nicht nur einen ersten Einblick in die ungewöhnliche Wechselwirkung zwischen einem Spiegel-Oligonukleotid und einem natürlichen Protein, sondern auch ein tieferes Verständnis der Funktionsweise beider Moleküle.“
Originalveröffentlichung
„Crystal structure of a mirror-image L-RNA aptamer (Spiegelmer) in complex with the natural L-protein target CCL2“; Dominik Oberthür et al.; „Nature Communications“, 2015
„Structural basis for the targeting of complement anaphylatoxin C5a using a mixed L-RNA/L-DNA aptamer“; Laure Yatime, Christian Maasch, Kai Hoehlig, Sven Klussmann, Gregers R. Andersen & Axel Vater; „Nature Communications“, 2015