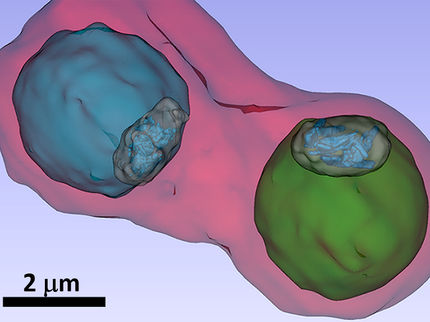
Das Innere einer Zelle aus Sicht eines sich bewegenden Proteins
Forscher entwickeln neue Methoden zur Bewegungsmessung von Proteinen in der Zelle
Zahlreiche Hindernisse, die sich aus den zellulären Strukturen ergeben, bremsen die Bewegungen von Proteinen innerhalb einer Zelle. Wissenschaftlern der Universität Heidelberg und des Deutschen Krebsforschungszentrums (DKFZ) ist es gelungen, die Zelltopologie zu „kartieren“, indem sie Proteine in der lebenden Zelle über verschiedene Zeitspannen und Distanzen beobachtet haben. Zum Einsatz kam dabei ein neues Verfahren, das auf Fluoreszenzmikroskopie basiert. Damit konnten die Wissenschaftler messen, wie lange Proteine brauchen, um im Zellinneren Strecken in einem Bereich von 0,2 bis 3 Mikrometern zurückzulegen. Aus der Analyse der Messdaten entwickelte das Team unter der Leitung von Dr. Karsten Rippe ein mathematisches Modell, mit dem sich intrazelluläre Strukturen rekonstruieren lassen. Die Forschungsergebnisse wurden in „Nature Communications“ veröffentlicht.
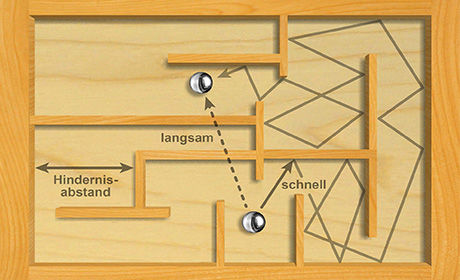
Die Bewegungen von Proteinen in der Zelle lassen sich mit denen einer Murmel in dem Spiel „Kugellabyrinth“ vergleichen: Auf kurzen Strecken bewegt sich die Murmel schnell, bis sie gegen ein Hindernis prallt. Das führt dazu, dass sie sich über längere Strecken mit einer verlangsamten Durchschnittsgeschwindigkeit per „stop and go“ fortbewegt. Indem die Wissenschaftler die insgesamt zurückgelegte Strecke und die dafür benötige Zeit kartierten, konnten sie den durchschnittlichen Abstand zwischen den Hindernissen messen und ein mathematisches Modell finden, mit dem sich die Proteinbewegung in der Zelle beschreiben lässt.
Karsten Rippe
Zellstrukturen wie Membranen, das Zytoskelett und das DNA-Genom bilden ein dynamisches dreidimensionales Labyrinth innerhalb der Zelle. Darin müssen sich Proteine zurechtfinden, um den Ort zu erreichen, an dem sie aktiv werden sollen. Die räumliche Struktur des Zellinneren ist deshalb ein wichtiger Faktor für den Transport von Proteinen und das Funktionieren der Zelle. „Zellstrukturen sind zwar in zahlreichen mikroskopischen Untersuchungen sichtbar gemacht worden. Bisher ist jedoch unklar, wie ein in der Zelle diffundierendes Protein dieses innere Netzwerk von Hindernissen ,erspürt‘“, sagt Dr. Rippe. Um dieser Frage nachzugehen, hat sein Team eine Methode entwickelt, mit der die räumliche Struktur in einer Zelle aus zufälligen Proteinbewegungen abgeleitet werden kann. Zum Einsatz kamen dabei fluoreszierende Proteine, die mit Hilfe eines selbst entwickelten Fluoreszenzspektroskopie-Systems beobachtet wurden. Wie Karsten Rippe erläutert, stellen dicht gepackte DNA-Bereiche im Zellkern die größten Hindernisse dar.
„Die Bewegungen eines Proteins in der Zelle ähneln denen einer Kugel in dem Geschicklichkeitsspiel, bei dem eine Murmel durch ein Labyrinth ,bugsiert‘ werden muss“, sagt Michael Baum, der Erstautor der Studie ist und die Arbeit als Teil seiner Dissertation an der Universität Heidelberg durchgeführt hat. Die Murmeln können leicht über kurze Strecken bewegt werden, dann kollidieren sie jedoch mit einem Hindernis und werden auf ihrem weiteren Weg gebremst. Das führt dazu, dass sie sich über längere Strecken mit einer verlangsamten Durchschnittsgeschwindigkeit per „stop and go“ fortbewegen. Bei der Analyse der Proteinbewegungen ordneten die Heidelberger Forscher der jeweiligen zurückgelegten Gesamtstrecke die dafür benötige Zeit zu. Daraus ergibt sich der durchschnittliche Abstand zwischen den Hindernissen. Auf diesen Daten basiert ein mathematisches Modell, mit dem sich die gemessene Bewegung der Proteine in der Zelle beschreiben und die Zelltopologie rekonstruieren lässt – und zwar mit einer deutlich besseren Auflösung als es aktuell mit einer lichtmikroskopischen Abbildung möglich ist, wie Dr. Rippe betont.
„Die Struktur der Hindernisse, auf die ein Protein bei seiner Bewegung durch die Zelle trifft, ähnelt dem Modell eines löchrigen Mediums, wie es zum Beispiel auch in einem Schwamm zu finden ist“, sagt der Heidelberger Wissenschaftler. In dieser dynamischen Struktur waren größere Proteine zuweilen mehrere Minuten lang gefangen. Ein weiteres Forschungsergebnis zeigt, dass sich Medikamente aus der Chemotherapie oder der Malaria-Behandlung ebenfalls auf die Mobilität der Proteine im Zellkern auswirken und die DNA-Hindernisse im Zellkern durchlässiger machen können. Dr. Rippe und sein Team wollen ihren neuen Ansatz nun in weiteren Untersuchungen am BioQuant-Zentrum der Universität Heidelberg und am DKFZ anwenden. Im Mittelpunkt stehen dabei die Wechselbeziehungen zwischen medikamentenbedingten Änderungen der Zellstruktur und dem Proteintransport sowie der Fehlregulierung dieses Prozesses bei Krankheiten.