Fischen im Proteinmeer
Zusammensetzung von Spleißkomplex in Chloroplasten erstmals identifiziert
Anzeigen
Um ein Gen in ein Protein zu übersetzen, bastelt eine Zelle zunächst einen Bauplan aus RNA. Einen der Hauptakteure in diesem Prozess haben Forscher um Dr. Jessica Jacobs von der Ruhr-Universität Bochum identifiziert. Das Team „fischte“ aus den Chloroplasten der Grünalge Chlamydomonas reinhardtii einen großen Komplex aus Proteinen und RNA, der am sogenannten Spleißen beteiligt ist. Er schneidet nicht-kodierende Bereiche aus der Boten-RNA, die den Proteinbauplan enthält. „Wir haben erstmals die exakte Zusammensetzung eines unbekannten Spleißkomplexes der Chloroplasten bestimmt“, sagt Jacobs.
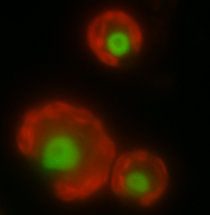
Fluoreszenzmikroskopische Aufnahme von Chlamydomonas reinhardtii. Der Zellkern der Alge ist durch ein grünleuchtendes Protein markiert, die Chloroplasten fluoreszieren rot.
© Allgemeine und Molekulare Botanik, RUB
Vom Gen zum Protein – Bastelarbeit gefragt
Gene, die Träger der Erbinformation, enthalten kodierende und nicht-kodierende Bereiche. Um ein Gen in ein Protein zu übersetzen, erstellen Enzyme zunächst eine Kopie des Gens, die Boten-RNA. Einen brauchbaren Bauplan für ein Protein erhält man aber nur, wenn Enzyme die nicht-kodierenden Bereiche, die sogenannten Intronen, aus der Boten-RNA herausschneiden. Diesen Prozess bezeichnen Wissenschaftler als Spleißen. Große Komplexe aus Proteinen und RNA sind für den Spleißvorgang verantwortlich.
Bestandteile des Spleißkomplexes in Chloroplasten identifiziert
Die RUB-Forscher untersuchten den Spleißvorgang des Gens psaA, das in Chloroplasten vorkommt. Diese Photosynthese treibenden Zellbestandteile von Pflanzen entstanden vermutlich aus ehemals freilebenden Cyanobakterien. Laut der Endosymbiontentheorie lebten die Cyanobakterien in Symbiose mit den Pflanzen und wurden schließlich in deren Zellen integriert. Daher besitzen Chloroplasten ein eigenes Erbgut – ein Relikt des Cyanobakterien-Erbguts. Allerdings sind die Chloroplasten auf die Kommunikation mit dem Zellkern angewiesen, um funktionsfähig zu sein. Das Bochumer Team identifizierte die Bestandteile des Proteinkomplexes, der das Chloroplasten-Gen psaA spleißt. In dem Spleißkomplex fanden sie 23 verschiedene Proteine, die im Erbgut des Zellkerns kodiert sind. „Der entdeckte Proteinkomplex ermöglicht uns einen Einblick in die Funktionsweise von Komponenten, die an der Kommunikation zwischen Chloroplasten und Zellkern beteiligt sind“, sagt Jessica Jacobs.
So angelt man einen Spleißkomplex
Die Untersuchungen führte das Team an der einzelligen Grünalge Chlamydomonas reinhardtii durch. „Wir haben ein Köderprotein genutzt, um den Spleißkomplex aus den Chloroplasten zu fischen“, erklärt Jacobs. Vor Beginn des Versuchs war bekannt, dass das Protein Raa4 am Spleißen des psaA-Gens beteiligt ist. Die vielen Interaktionspartner von Raa4 waren jedoch unbekannt. Die RUB-Biologen veränderten die Grünalge genetisch so, dass sie eine modifizierte Form des Raa4-Proteins herstellte – mit „Tag“, also mit einer Art „Angelhaken“. Sie isolierten alle Proteine der Zelle und filterten sie durch ein bestimmtes Material, an dem nur Raa4 mit Angelhaken hängen blieb – inklusive all seiner gebundenen Interaktionspartner. Die Bestandteile des herausgefischten Spleißkomplexes bestimmten sie mit der Massenspektrometrie. Für verschiedene Umweltbedingungen fanden die Forscher einen gleich zusammengesetzten Spleißkomplex: bei Licht, Dunkelheit sowie in sauerstofffreier Umgebung.
Originalveröffentlichung
Weitere News aus dem Ressort Wissenschaft
Meistgelesene News
Weitere News von unseren anderen Portalen
Verwandte Inhalte finden Sie in den Themenwelten
Themenwelt Massenspektrometrie
Die Massenspektrometrie ermöglicht es uns, Moleküle aufzuspüren, zu identifizieren und ihre Struktur zu enthüllen. Ob in der Chemie, Biochemie oder Forensik – Massenspektrometrie eröffnet uns ungeahnte Einblicke in die Zusammensetzung unserer Welt. Tauchen Sie ein in die faszinierende Welt der Massenspektrometrie!
Themenwelt Massenspektrometrie
Die Massenspektrometrie ermöglicht es uns, Moleküle aufzuspüren, zu identifizieren und ihre Struktur zu enthüllen. Ob in der Chemie, Biochemie oder Forensik – Massenspektrometrie eröffnet uns ungeahnte Einblicke in die Zusammensetzung unserer Welt. Tauchen Sie ein in die faszinierende Welt der Massenspektrometrie!