Kleine RNA als Baukasten
Stress gibt es auch bei krank machenden Bakterien - etwa wenn sie vom Immunsystem ihres Wirtes attackiert werden. Wie Salmonellen auf eine solche Belastung reagieren, beschreiben Würzburger Infektionsbiologen in der Zeitschrift PNAS.
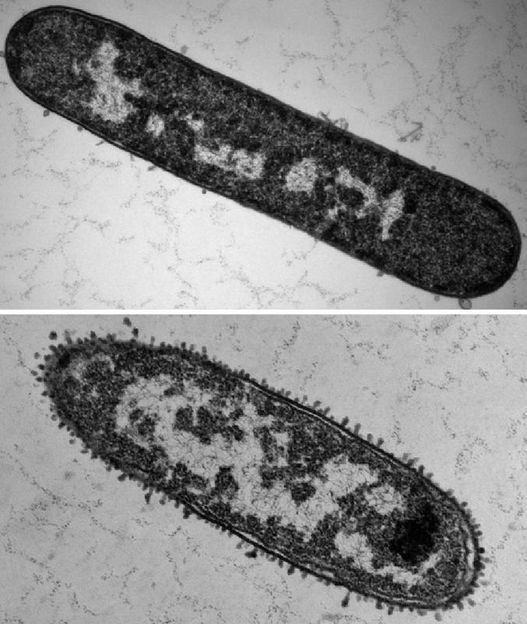
Zwei Salmonellen:Das obere Bakterium ist unversehrt, das untere wurde mit antimikrobiellen Peptiden behandelt. Seine Außenhülle zerfällt, Material aus dem Zellinneren läuft in die Umgebung.
Institut für Molekulare Infektionsbiologie
Salmonellen sind Bakterien, die beim Menschen heftigen Durchfall auslösen können. Wenn sie sich im Verdauungstrakt breit machen, bleiben sie nicht unbehelligt: Das Immunsystem greift die Eindringlinge unter anderem mit Peptiden an. Das sind kleine Eiweißkörper, die Löcher in die äußere Hülle der Bakterien reißen.
Sobald ihre Hülle beschädigt ist, reagieren die Salmonellen: Unter anderem produzieren sie dann ein kleines RNA-Molekül (RybB-sRNA), das in der Bakterienzelle schlagartig die Synthese von etwa zehn Proteinen unterbindet. Dabei handelt es sich allesamt um Proteine, die für einen Einsatz in der Außenhülle der Bakterien vorgesehen sind.
Ein sinnvoller Mechanismus: „Die Salmonellen helfen sich damit ganz schnell. Weil ihre äußere Membran löchrig ist, würden die Proteine dort keinen Halt finden und nicht funktionieren“, erklärt Kai Papenfort vom Institut für Molekulare Infektionsbiologie der Universität Würzburg. Letzten Endes verhindert das kleine RNA-Molekül also eine Verschwendung von Protein-Ressourcen.
RNA-Anfang bindet Vorstufen der Proteine
Wie aber schafft es die kleine RNA, auf einen Schlag die Produktion gleich mehrerer Proteine zu regulieren? Diese Frage beantworten die Würzburger Forscher in der aktuellen Ausgabe der Zeitschrift PNAS: „Der Anfangsbereich des sRNA-Moleküls bindet die Transkripte, das ist so eine Art Vorstufe all dieser Proteine“, sagt Institutsleiter Professor Jörg Vogel. „Sobald das geschehen ist, stoppt die Produktion.“ Zum Beweis übertrugen die Forscher den Anfangsbereich auf andere RNA-Moleküle. Auch diese brachten daraufhin die Herstellung der zehn Proteine ins Stocken.
In der Evolution der Bakterien nicht verändert
Erstmals haben die Würzburger Forscher damit gezeigt: Auch kleine RNA-Moleküle besitzen klar abgrenzbare Bereiche, denen sich eine regulierende Funktion zuweisen lässt. Bislang war das nur für Proteine bekannt, nicht aber für „einfachere“ Moleküle wie RNA. „Auch RNA besteht aus funktionellen Stücken, die sich nach dem Baukastenprinzip neu anordnen lassen“, so Vogel.
Bei dem regulierenden Bereich handelt es sich zudem um ein Stück RNA, das sich bei der Evolution der Bakterien nicht verändert hat. Das heißt: „Diese RNA gibt es nicht nur bei Salmonellen, sondern auch bei vielen anderen krankheitserregenden Bakterien, und sie übt immer dieselbe Funktion aus“, so Kai Papenfort.
Eine molekulare Struktur, die in der Evolution gleich geblieben ist - das deutet darauf hin, dass es sich um etwas sehr Wichtiges handelt. Um einen Faktor womöglich, den Bakterien beim Infektionsprozess unbedingt brauchen. Der möglicherweise beim Auslösen der Krankheit eine Rolle spielt. Ob das so ist, wollen die Würzburger Forscher als nächstes klären. Am Ende könnte sich der Anfangsbereich der RybB-sRNA als potenzieller Angriffspunkt für neue Medikamente erweisen.
Grundlagenforschung über kleine RNA
Das Team von Professor Jörg Vogel betreibt Grundlagenforschung über kleine RNA-Moleküle, deren Ketten aus ungefähr 100 Bausteinen bestehen (small RNA, kurz: sRNA). Dieser spezielle RNA-Typus reguliert Lebensvorgänge in Bakterien und höher entwickelten Zellen. Als Modellorganismen benutzen die Würzburger Wissenschaftler neben Salmonellen auch Helicobacter, ein Bakterium, das Magenkrebs auslösen kann.
Originalveröffentlichung: „Evidence for an autonomous 5‘ target recognition domain in an Hfq-associated small RNA”, Kai Papenfort, Marie Bouvier, Franziska Mika, Cynthia M. Sharma, and Jörg Vogel; PNAS, online publiziert am 8. November 2010


























































