Enzyme aus einem Guss: CRISPR-Cas-Komponenten arbeiten zusammen, um den Schutz vor Viren zu verbessern
Anzeigen
Forscher von Skoltech und ihre Kollegen aus Russland und den USA haben gezeigt, dass die beiden Komponenten des bakteriellen CRISPR-Cas-Immunsystems - eine, die fremde genetische Elemente wie Viren zerstört, und eine andere, die "Erinnerungen" an fremde genetische Elemente schafft, indem sie Fragmente ihrer DNA an einer speziellen Stelle des bakteriellen Genoms speichert - physikalisch miteinander verbunden sind. Diese Verbindung hilft Bakterien, ihr Immungedächtnis effizient zu aktualisieren, wenn sie von mutierten Viren infiziert werden, die gelernt haben, der CRISPR-Cas-Abwehr zu entgehen. Die Arbeit wurde in der Zeitschrift Proceedings of the National Academy of Sciences veröffentlicht.
Pavel Odinev / Skoltech
CRISPR-Cas, ein Abwehrmechanismus, der Bakterien eine Resistenz gegen ihre Viren (Bakteriophagen) verleiht, zerstört die DNA von zuvor angetroffenen Gegnern, indem er sie mit Spacern "vergleicht", kurzen Bits genetischer Information, die als "Bibliothek" in einem "Speicherchip" gespeichert sind, der sich an einer speziellen Stelle des bakteriellen Genoms befindet. CRISPR-Cas "lernt", neue Gegner zu erkennen, indem es während der Infektion neue, von Viren stammende Spacer in diese Bibliothek einbaut. Die beiden Phasen der CRISPR-Funktion, der Erwerb von Spacern und deren Verwendung zur Bekämpfung von Reinfektionen, werden entsprechend als Adaptation und Interferenz bezeichnet.
"Um CRISPR zu umgehen, erwerben die Phagen Mutationen, die Fehlpaarungen mit Spacern einführen. Um eine effektive Verteidigung aufrechtzuerhalten, muss das CRISPR-Cas-System also den Satz der Spacer schneller aktualisieren, als mutierte Phagen mit Escape-Mutationen entstehen. Um diese Anforderung zu erfüllen, haben CRISPR-Cas-Systeme einen speziellen Mechanismus der "primed adaptation" entwickelt. Während der Primed Adaptation fördern bereits existierende Spacer, die ein Ziel erkennen, auch wenn sie ineffizient sind, eine sehr effiziente Akquisition zusätzlicher Spacer von demselben DNA-Molekül, auf dem sich das Ziel befindet", erklärt Olga Musharova, Skoltech-Forscherin und Erstautorin der Arbeit.
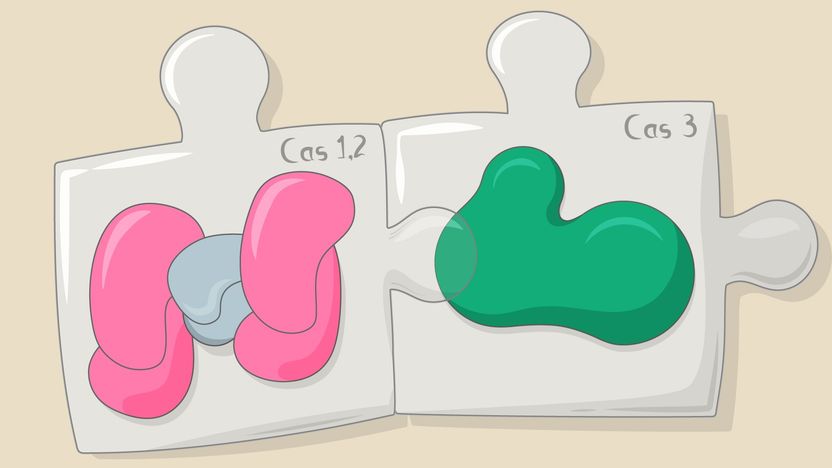
Der genaue molekulare Mechanismus des Primings ist noch unklar, aber er erfordert offensichtlich eine enge Koordination zwischen dem tötenden und dem speichernden Teil des CRISPR-Mechanismus. In der neuen Arbeit konnten Skoltech-Professor Konstantin Severinov, Musharova und ihre Kollegen die Existenz eines "Priming-Komplexes" bestätigen, der sowohl die Cas1-Cas2-Proteine umfasst, die für die Aufnahme neuer Spacer verantwortlich sind, als auch das Cas3-Protein, das die feindliche DNA spaltet.
"Der Teil, der die fremde DNA zerstört, und der Teil, der neue Informationen für die zukünftige Schutzfunktion des CRISPR-Cas-Systems erwirbt, sind miteinander verbunden. Das ist so, als ob der Schläger in einem Whac-A-Mole-Spiel auch Bilder von den Maulwürfen als zukünftige Referenz aufnehmen könnte", sagte Severinov.
In Experimenten mit E. coli zeigte das Team, dass DNA-Fragmente, die von Cas3 abgebaut werden, direkt an Cas1-Cas2 als "Prespacer" weitergegeben werden, die schließlich zu Spacern werden. "Dieses Ergebnis ist von fundamentaler Bedeutung. Wir haben die Verbindung zwischen der Interferenz und den Anpassungsprozessen aufgedeckt", sagt Musharova. "Unsere Ergebnisse zeigen auch, wie die CRISPR-Anpassung effizienter gestaltet werden kann, was für die Nutzung von Bakterienpopulationen zur Informationsspeicherung wichtig ist."
Das Team plant, die grundierte Anpassung in Bakterienzellen weiter zu untersuchen und den effizientesten Weg zu finden, um gewünschte "Erinnerungen" in Form von Spacern in die bakterielle DNA einzubauen.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.