Mit Inhibitoren gegen das SARS-CoV-2-Enzym vorgehen
Das COVID-19-Virus hat eine Achillesferse, was es zu einem ausgezeichneten Ziel für Hemmstoffe oder neue Medikamente macht
Anzeigen
Während sich die durch das Coronavirus SARS-CoV-2 verursachte Pandemie Covid-19 weiterhin weltweit ausbreitet, untersuchen viele Forscher epidemiologische Modelle, um ihre Ausbreitung vorherzusagen.
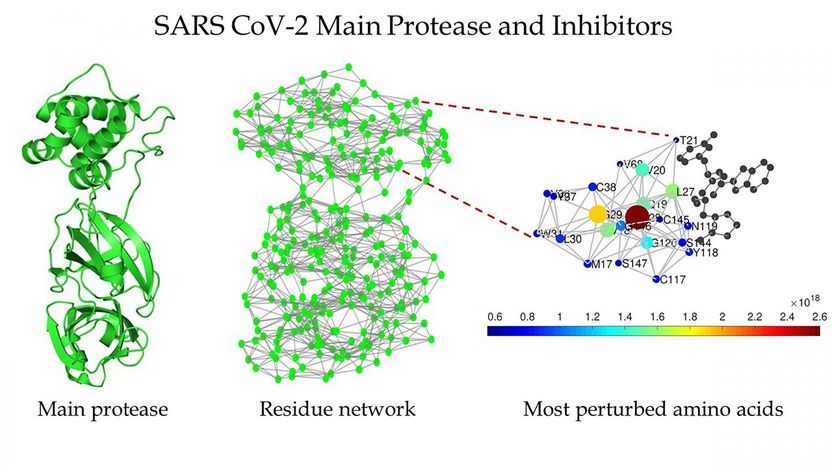
Schematische Darstellung der Hauptprotease von SARS CoV-2 (links), des Proteinrest-Netzwerks der Hauptprotease von SARS CoV-2 (Mitte) und eine vergrößerte Ansicht der Region um die Bindungsstelle, wie sie von Estrada detektiert wird (rechts).
Ernesto Estrada
Ernesto Estrada, ein Mathematiker und Experte für komplexe Systeme der Stiftung ARAID an der University of Zaragoza, beschloss jedoch, sich darauf zu konzentrieren, innerhalb von SARS-CoV-2 Angriffsziele für neue Medikamente zu finden. Aus früheren Arbeiten wusste er, dass die Haupt-Protease des Virus, ein Enzym, das für die proteolytische Verarbeitung von Polyproteinen zuständig ist, ein hervorragendes Ziel ist.
In der Zeitschrift Chaos, von AIP Publishing, sagte Estrada, als er und seine Kollegen einen dramatischen Anstieg der Empfindlichkeit der Haupt-Protease von SARS-CoV-2 gegenüber kleinen Störungen entdeckten, machte es sie misstrauisch, dass Inhibitoren eine Rolle bei der Abtötung des Virus spielen könnten.
Inhibitoren sind organische Moleküle, Medikamente oder neue chemische Verbindungen, die sich an die Bindungsstelle einer Protease anlagern, um ihre Arbeit zu hemmen. Ein Virus stirbt, ohne dass ein proteolytisches Enzym für es arbeitet.
"Mir fiel auf, dass Chemiker bereits einige potente Inhibitoren der Hauptprotease von SARS-CoV-2 gefunden hatten, und dass sie die Struktur dieses Proteins mittels Röntgenkristallographie aufgelöst hatten", sagte er. "Es war schockierend zu sehen, dass diese Protease der des SARS-Coronavirus sehr ähnlich ist, das die Epidemien des Jahres 2003, SARS-CoV-1, verursachte.
Als die Forscher beide Strukturen übereinander legten, passten sie fast perfekt zusammen.
"Wenn man die Aminosäuresequenzen beider Proteasen aneinanderreiht, gibt es nur 12 von 306 Resten, die nicht übereinstimmen", sagte Estrada. "Verbirgt sich etwas hinter diesen scheinbaren Ähnlichkeiten zwischen den beiden Proteasen? Können wir etwas von ihnen lernen, um das Design von Medikamenten gegen das Virus zu verbessern?
Estradas Gruppe hat umfangreiche Erfahrung in der Analyse von Netzwerken - wie sozialen Netzwerken, dem Internet oder Nahrungsketten zwischen Arten innerhalb einer Umgebung - und beschloss, ein Protein als Netzwerk zu behandeln.
"Man nennt sie Proteinrest-Netzwerke, in denen wir jede Aminosäure als Knotenpunkt darstellen, und die Interaktion zwischen zwei Aminosäuren wird durch eine Verbindung zwischen den beiden dargestellt", erklärte er.
Sie fanden mehrere Strukturen der Hauptprotease von SARS-CoV-1 und SARS-CoV-2, die sauber waren, was bedeutet, dass sie keine Mutationen, Liganden oder Lösungsmittel in ihren Strukturen enthalten. Sie wandelten ihre Struktur in Proteinrest-Netzwerke um.
Estrada sagte, dass die meisten traditionellen Netzwerk-Messungen zeigten, dass beide Strukturen einander sehr ähnlich seien, was sein Team bereits wusste. "Aber vor ein paar Jahren haben wir ein ausgefeilteres mathematisches Maß entwickelt, mit dem wir feststellen können, wie weit sich eine Störung innerhalb eines Netzwerks ausbreiten kann. Diese Arbeit war von sehr theoretischer, mathematischer Natur, aber wir hatten spekuliert, dass sie für die Untersuchung von Proteinen nützlich sein könnte.
Also haben sie es auf den Prüfstand gestellt. Es zeigte sich, dass die Protease von SARS-CoV-2 um 1.900% empfindlicher auf die Fernübertragung von Störungen reagiert als die Protease von SARS-CoV-1.
"Das bedeutet, dass, wenn ein Protein gestört wird, zum Beispiel durch Wasser in der intrazellulären Umgebung, solche Störungen durch ein Netzwerk von Intraresiduen übertragen werden, die die 3D-Struktur des Proteins bilden", sagte Estrada. "Wenn eine solche Störung um eine bestimmte Aminosäure innerhalb der Protease von SARS-CoV-1 herum erzeugt wird, wird sie nur durch eine enge Umgebung um diese gestörte Aminosäure herum übertragen.
Wenn diese Störung jedoch an einer Aminosäure innerhalb der Protease von SARS-CoV-2 auftritt, wird sie auf fast das gesamte Netzwerk übertragen - sogar auf Aminosäuren, die sehr weit entfernt sind.
"Das ist bemerkenswert, denn es bedeutet, dass bei winzigen strukturellen Unterschieden die Protease von SARS-CoV-2 innerhalb der Intrareside-Kommunikation viel effektiver ist", sagte Estrada. "Sie sollte ihre Aufgabe als proteolytisches Enzym des Virus viel effektiver erfüllen. Der Teufel hat hier eine fast perfekte Arbeit geleistet, aber er hat die Tür offen gelassen. Diese große Empfindlichkeit der SARS-CoV-2-Protease gegenüber Störungen kann ihre Achillesferse gegenüber Inhibitoren sein".
Der Ansatz der Gruppe kann für massive Screening-Protokolle zur Identifizierung potenter Inhibitoren der SARS-CoV-2-Hauptprotease und folglich für die Entwicklung neuer Medikamente zu ihrer Abtötung verwendet werden.
Hinweis: Dieser Artikel wurde mit einem Computersystem ohne menschlichen Eingriff übersetzt. LUMITOS bietet diese automatischen Übersetzungen an, um eine größere Bandbreite an aktuellen Nachrichten zu präsentieren. Da dieser Artikel mit automatischer Übersetzung übersetzt wurde, ist es möglich, dass er Fehler im Vokabular, in der Syntax oder in der Grammatik enthält. Den ursprünglichen Artikel in Englisch finden Sie hier.