Metastasierung von Tumoren verhindern
Paul Scherrer Institut und Roche legen Grundstein für Entwicklung eines Medikaments gegen Metastasierung
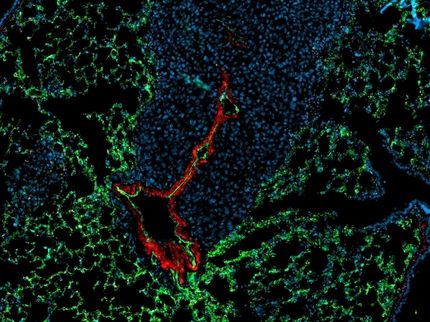
Wenn Krebszellen sich im Körper ausbreiten, können Tochtergeschwülste, sogenannte Metastasen, entstehen. Diese sind für etwa 90 Prozent der Todesfälle bei Krebspatienten verantwortlich. Ein wichtiger Ausbreitungsweg der Krebszellen verläuft über das Lymphgefässsystem, das, ähnlich wie das Blutgefässsystem, den ganzen Körper durchzieht und Lymphknoten miteinander verbindet. Bei der Wanderung von weißen Blutzellen durch dieses System, um beispielsweise die Abwehr von Krankheitserregern zu koordinieren, spielt ein spezielles Membranprotein, der Chemokin-Rezeptor 7 (CCR7), eine wichtige Rolle. Dieser sitzt in der Hülle der Zellen, der Zellmembran, und zwar so, dass er äussere Signale empfangen und diese in das Innere weiterleiten kann. Im Rahmen eines gemeinsamen Projekts mit dem Pharmaunternehmen F. Hoffmann-La Roche AG (Roche) haben Forscher des Paul Scherrer Instituts PSI erstmals die Struktur von CCR7 entschlüsseln und den Grundstein für die Entwicklung eines Medikaments legen können, das die Metastasierung bestimmter häufiger Krebsarten wie Darmkrebs verhindern könnte.

Steffen Brünle (rechts) und Jörg Standfuss an der Apparatur, mit der sie Proteine voneinander trennen. Für ihre Studie veränderten die Forschenden Insektenzellen so, dass sie ein menschliches Protein herstellen. Um dieses aus der Zelle zu extrahieren, wurde die Zelle zerstört und mithilfe dieser Apparatur dann das Protein separiert, dessen Struktur die Forschenden aufgeklärt haben.
Paul Scherrer Institut/Markus Fischer
In den Zellen aller Wirbeltiere kommen 20 verschiedene Chemokin-Rezeptoren vor, die mit mehr als 40 Signalproteinen, sogenannten Chemokinen, interagieren können. Jedes dieser Signalproteine passt nur zu ganz speziellen Rezeptoren. Bindet eines der Signalproteine an einen Rezeptor, löst das wiederum Prozesse innerhalb der Zelle aus, die zu einer spezifischen zellulären Antwort auf das Signal führt.
Der Rezeptor CCR7 ist einer der Rezeptoren, der die Bewegung von Zellen im Körper steuert. Sobald das passende Signalprotein ausserhalb der Zelle an ihn bindet, führt eine Kettenreaktion in der Zelle dazu, dass sich die Zelle in Richtung der höchsten Konzentration des Signalproteins bewegt. Die Zelle folgt der Spur der Chemokine quasi wie ein Spürhund. So wird beispielsweise ein konstanter Fluss von weissen Blutkörperchen, wichtigen Zellen des Immunsystems des Körpers, zu den Lymphknoten gesteuert.
Auch Krebszellen können sich CCR7 zu Nutze machen und den Zellrezeptor so für ihre Zwecke missbrauchen. Das passende Signalprotein leitet ihnen den Weg aus dem Tumor heraus und in das Lymphgefässsystem hinein. Darüber breiten sie sich im Körper aus und bilden in anderen Geweben schliesslich Metastasen. Diese Tochtergeschwulste erhöhen die Sterblichkeit der Betroffenen drastisch.
Künstliche Wirkstoffe verhindern die Zellwanderung
Um die Überlebensrate von Krebspatienten zu erhöhen, ist es von grossem medizinischem Interesse den Prozess der Metastasierung zu unterbinden. PSI-Forscher haben deshalb mithilfe von Röntgenkristallografie an der Synchrotron Lichtquelle Schweiz SLS am PSI die Struktur des CCR7-Rezeptors entschlüsselt. Diese Struktur diente in einer Zusammenarbeit mit Roche als Grundlage für die Suche nach entsprechenden Wirkstoffen. «Das passende Molekül kann verhindern, dass das Signalprotein an den Rezeptor koppelt und in der Zelle zu einer Reaktion führt», erklärt Steffen Brünle, der als PSI-FELLOW-II-3i und einer der beiden Erstautoren die Studie durchführte. Die Struktur des Rezeptors zu entschlüsseln war eine echte Herausforderung. «Das Schwierige daran war, die Proteine überhaupt so herzustellen, dass wir sie röntgenkristallografisch untersuchen konnten», so Jörg Standfuss, Co-Leiter des Projekts und der Arbeitsgruppe Zeitaufgelöste Kristallografie am PSI. Um den Untersuchungsprozess zu beschleunigen entwickelte Roche eigens neue proteinverändernde Technologiebausteine, sogenannte Kristallisations-Chaperone.
Mit den Informationen über die genaue Struktur des Rezeptors konnten sie ein passendes Molekül identifizieren, das den Rezeptor blockiert und somit verhindert, dass ein Signal in die Zelle weitergegeben wird. «Unsere Experimente zeigen, dass das künstliche Molekül im Inneren der Zelle an den Rezeptor bindet. Dadurch wird verhindert, dass die Kettenreaktion, die zur Zellwanderung führt, gestartet wird», so Brünle.
Der Wert wissenschaftlicher Zusammenarbeit
Aus Millionen von Molekülen, die bei Roche in einer Datenbank deponiert sind, und mithilfe der Struktur des Wirkstoff-gebundenen Rezeptors, haben die Roche-Wissenschaftler mittels Computersimulation nach den passenden Wirkstoffen gesucht, die sich zum Blockieren des Signalproteins eignen könnten, und identifizierten fünf Substanzen als mögliche Kandidaten zur Weiterentwicklung potenzieller Wirkstoffe in der Krebstherapie.
Einer der Wirkstoffe, den die Forscher in ihrer Studie entdeckten, wird auch schon in der Pharmaindustrie als potenzielles Medikament gegen Metastasierung in klinischen Studien getestet. Allerdings ging man bisher davon aus, dass er an einen anderen Rezeptor bindet, und daher eine andere Funktion der Krebszelle hemmt. Für die Pharma-Forschung und -entwicklung sind neue Erkenntnisse aus Studien wie dieser daher ausserordentlich wertvoll.
Originalveröffentlichung
"Structural basis for allosteric ligand recognition in the human CC chemokine receptor 7"; K. Jaeger, S. Bruenle, T. Weinert, W. Guba, J. Muehle1, T. Miyazaki, M. Weber, A. Furrer, N. Haenggi, T. Tetaz, C. Huang, D. Mattle, J.-M. Vonach, A. Gast, A. Kuglstatter, M.G. Rudolph, P. Nogly, J. Benz, R.J.P. Dawson, J. Standfuss; Cell; 22. August 2019 (online)